Estudio de detección molecular del Virus de la Leucemia Felina (FeLV) y análisis filogenético en gatos domésticos de Tenerife, España.
Resumen breve
El objetivo de este trabajo fuer desarrollar una herramienta molecular capaz de detectar FeLV en gatos domésticos infectados naturalmente, y realizar una clasificación filogenética de las secuencias obtenidas en el estudio. Este trabajo proporciona el primer informe molecular de FeLV realizado en Tenerife en el que, además, se detecta una alta prevalencia, destacando la importancia de realizar más estudios en la isla para comprender mejor el alcance de esta infección.Índice de contenidos

Resumen
El virus de la leucemia felina (FeLV) es un retrovirus asociado con múltiples síndromes de enfermedades mortales que afectan gravemente la salud de los felinos, desde linces y panteras salvajes hasta gatos domésticos. Su prevalencia es mundial, pero en Tenerife no hay información sobre su situación en la población de gatos domésticos. Además, la detección molecular generalmente se ve obstaculizada ya que el virus comparte homología con secuencias endógenas de infecciones antiguas de FeLV, lo que hace que sea un desafío. El objetivo de este trabajo fuer desarrollar una herramienta molecular capaz de detectar FeLV en gatos domésticos infectados naturalmente, y realizar una clasificación filogenética de las secuencias obtenidas en el estudio. Para ello, se desarrolló una reacción en cadena de la polimerasa (PCR) para amplificar el gen env del provirus de ADN y tras secuenciar las muestras, se analizaron las relaciones filogenéticas entre las secuencias obtenidas y un grupo de secuencias representativo de los subtipos de FeLV obtenidos de la base de datos GenBank. Se obtuvo un 30.2 % de muestras positivas, siendo FeLV-A el subtipo predominante, con algunas secuencias estrechamente relacionadas con las variantes Arena, Glasgow-1 y FeLV-FAIDS. Las cepas de FeLV detectadas están altamente conservadas y estrechamente relacionadas entre sí. Este trabajo proporciona el primer informe molecular de FeLV realizado en Tenerife en el que, además, se detecta una alta prevalencia, destacando la importancia de realizar más estudios en la isla para comprender mejor el alcance de esta infección.
Palabras clave: Leucemia felina (FeLV), PCR, diagnóstico, filogenia
Introducción
El virus de la leucemia felina (FeLV) es un virus de ARN monocatenario de sentido positivo de la familia Retroviridae, capaz de provocar síndromes de enfermedades fatales en felinos, incluyendo neoplasias y linfoma, así como enfermedades degenerativas del sistema hematopoyético y anemia aplásica1. Este virus es responsable de más muertes entre los felinos que cualquier otra enfermedad infecciosa, afectando tanto a los gatos domésticos como a felinos salvajes en todo el mundo2,3. Su relevancia radica, no solo en la gravedad de sus manifestaciones clínicas, sino también en la facilidad con la que se propaga. Si el sistema inmunológico del gato no neutraliza el virus después de la infección inicial, el virus se disemina hasta la médula ósea causando trastornos, infectando las células madre precursoras del sistema hematopoyético, y en la mayoría de los gatos infectados con FeLV, aparece una anemia no neoplásica mediada por el sistema inmunológico como uno de sus muchos resultados2. Las tasas de mortalidad pueden alcanzar el 50 % seis meses después de la infección, y hasta el 80 % dentro de los 3 años de la infección4.
La relación entre el genoma del gato y FeLV presenta muchas singularidades de la familia de los retrovirus, incluyendo la aparición de diferentes subgrupos debido a eventos de recombinación genética durante la integración del provirus en el genoma del gato5–7. Como contrapartida del virus exógeno, integrado en el genoma de cada línea celular de la mayoría de los gatos domésticos, están presentes elementos endógenos conocidos como secuencias enFeLV, con una estimación de 6 a 12 copias por genoma haploide, que representan huellas de infecciones retrovirales antiguas que se transmiten de padres a hijos8,9. A pesar de no formar partículas infecciosas ni ser capaces de inducir la enfermedad por sí mismos, su presencia impacta significativamente en el diseño de un ensayo de detección molecular, ya que su homología con una secuencia de FeLV exógeno podría alcanzar un 86 %. Si estos elementos no funcionales están presentes durante una infección natural por FeLV, la región homóloga del FeLV endógeno (enFeLV) puede recombinarse con el virus exógeno (exFeLV) durante la transcripción inversa viral y la integración, generando nuevos subgrupos recombinantes5,10. Existe una clasificación de subgrupos de FeLV basada en manifestaciones funcionales y fenotípicas, sin embargo, en este estudio nos centramos en los más frecuentes: FeLV-A, que se encuentra presente en todos los gatos infectados con FeLV y causa una grave inmunosupresión, anemia macrocítica y linfoma7; y FeLV-B, una forma recombinante de FeLV-A con elementos de FeLV endógeno (enFeLV), que ocurre en aproximadamente el 50 % de las infecciones, y causa enfermedades neoplásicas como tumores y otros crecimientos anormales del tejido2,7. Además, existe un subgrupo FeLV-C, pero solo se observa en el 1 % de los gatos infectados con FeLV, que causa anemia severa11.
La prevención de la mortalidad no es posible, y aunque la quimioterapia puede retrasarla, el diagnóstico temprano es crucial para reducir el ciclo de transmisión y gestionar los resultados de la infección. Hay tres etapas de la infección por FeLV y cada una tiene sus propias características, lo que permite la detección mediante un test de antígeno p27 o por PCR (proviral o ARN)12. Durante la etapa progresiva de la infección, la viremia alcanza su pico máximo 2-4 semanas después de la infección, y los gatos se convierten en hospedadores contagiosos. El ARN de FeLV se convierte en provirus de ADN exógeno al integrarse en el genoma del gato mientras el virus se propaga en la circulación sanguínea. En este punto, una prueba de antígeno p27 podría ser el método tradicional de detección, aunque puede dar falsos negativos en las etapas tempranas de viremia, como sucede con otras pruebas inmunocromatográficas rápidas habitualmente utilizadas4. Si el sistema inmunológico del gato logra combatir la infección temprana, entonces el provirus de ADN no se integraría y no podría ser detectado por ningún tipo de prueba después de cierto tiempo. Este sería el caso de una infección abortiva, caracterizada por la ausencia de antígeno y del ADN del provirus de FeLV ya sea en la circulación o en los tejidos, debido a que el animal ha logrado eliminar la infección. De manera similar a la etapa abortiva, en la etapa regresiva, la antigenemia es indetectable13. Sin embargo, el ARN de FeLV se integrará como provirus de ADN, convirtiendo al gato en un hospedador capaz de transmitir verticalmente la infección y, bajo condiciones de inmunosupresión, también de manera horizontal, acompañado con el desarrollo de las enfermedades asociadas a FeLV4,12,14,15. Incluso, podría ocurrir la supresión de la médula ósea o la aparición de linfoma en gatos regresivos sin la reactivación de la infección15. En consecuencia, la cinética de la infección por FeLV despliega una batalla entre el virus y el sistema inmunológico, no solo durante la infección inicial, sino también, en algunos casos, a lo largo de toda la vida.
La prevalencia de FeLV es amplia en diferentes áreas geográficas y hasta la fecha solo unos pocos estudios han examinado su impacto en España. Algunos de ellos realizados en Cataluña informaron de tasas de prevalencia que oscilan entre el 2 y el 8% de la población felina, incluyendo a los gatos callejeros16–18. Este porcentaje puede aumentar hasta un 13 % dependiendo de las condiciones del trabajo, como las reportadas por Arjona y Ravicini17,18 para Madrid y Barcelona. Las diferencias entre estos ensayos estaban asociadas al origen de los animales, que procedían de refugios o eran gatos callejeros, que presentan tasas de detección aumentadas, ya que estas clases de gatos suelen estar significativamente más expuestas al virus. Tenerife es la isla más grande del archipiélago de las Islas Canarias, ubicada al oeste del continente africano, y alberga una gran población de gatos domésticos y callejeros, así como en refugios. Sin embargo, no hay información sobre la situación actual por FeLV, su prevalencia y filogenia.
Actualmente es un hecho que hay una disminución en la prevalencia del FeLV como resultado de la implementación de programas de vacunación y pruebas1. Sin embargo, estudios recientes indican que el progreso se ha estancado en numerosos países. Por lo tanto, es importante actualizar la información de manera constante para prevenir la incidencia de la enfermedad12,15,19,20. El uso de la vacuna contra el FeLV no proporciona protección completa, sin embargo, tiene la capacidad de reducir el impacto de la infección en gatos no expuestos al prevenir la viremia persistente, aunque no previene la integración del provirus de ADN tras la exposición12. A su vez, el estado vacunal de los gatos no interfiere con la detección molecular, por lo que el testeo debe realizarse periódicamente para asegurarse que no ha ocurrido la infección tiempo después de la vacunación. Y si bien las vacunas actuales han logrado reducir significativamente la prevalencia de FeLV donde se lleva a cabo la vacunación rutinaria, la búsqueda de nuevas vacunas mejoradas debe continuar1–3.
Con el tiempo, la elección por excelencia para la detección del provirus de FeLV han sido los métodos basados en PCR, en lugar de las pruebas de detección de antígenos y las pruebas de interferencia viral (IAs)4,21. Sin embargo, la presencia de secuencias enFeLV en la mayoría de los gatos domésticos ha añadido desafíos adicionales a los ensayos de detección molecular. Para distinguir entre las secuencias enFeLV y exFeLV, las regiones dentro del gen env (gen de la envoltura) han sido las más útiles para desarrollar cebadores específicos para PCR5,10,21,22. Además, están surgiendo nuevos métodos basados en técnicas moleculares, más específicamente en la reconstrucción filogenética del gen SU (Surface, en inglés)5,19,20,22 usando las diferencias presentes en esa región que permiten la amplificación diferencial por PCR y la caracterización de los subgrupos de FeLV6,23, analizando la variación entre FeLV-A y enFeLV principalmente en las regiones LTR y env5,19,24. En consecuencia, el objetivo de este estudio fue desarrollar un ensayo de PCR con fines diagnósticos y filogenéticos, para explorar una visión inicial de la prevalencia del FeLV en Tenerife en gatos domésticos naturalmente infectados y sus asociaciones filogenéticas.
Materiales and Métodos
Selección de casos y recolección de datos
Se analizaron un total de 96 muestras entre 2018 y 2022, recogidas de gatos llevados a clínicas veterinarias para chequeos rutinarios o por presentar signos clínicos. Dado que no se proporcionó información detallada sobre el historial clínico, la selección de casos se basó únicamente en la elección de los propietarios de testar a sus mascotas. Se analizaron variables como la edad, raza, sexo y área de la clínica veterinaria de los casos disponibles. Las muestras fueron recolectadas por personal veterinario cualificado en las clínicas, siguiendo los procedimientos éticos estándar.
Procesamiento de muestras y extracción de ácidos nucleicos
Se recogieron muestras de sangre periférica en tubos con EDTA y se enviaron para su análisis al Laboratorio Finca España, Tenerife, España. Después de la recolección, se realizó la extracción de ADN utilizando el kit de NucleoSpin® (Macherey-Nagel, Alemania) siguiendo las recomendaciones del fabricante. La cuantificación de ADN de las muestras se llevó a cabo en un Nanodrop ND-1000® (Thermo Scientific) y se almacenaron a -20 °C hasta su procesado.
Diseño de cebadoresy optimización de la PCR
La región de interés fue seleccionada para garantizar la detección específica del gen env del provirus de ADN de FeLV 21. Los cebadores de PCR fueron diseñados utilizando el programa GeneRunner® y sus secuencias son: FWD: 5′-AAA CCA ACA CCC AAG CTA ATG CCA -3′ y REV: 5′-GGC AGA CGT AAA AGG GGTATG T-3′, amplificando un fragmento de 217 pares de bases. Los cebadores presentaron una homología del 100 % con secuencias de referencia de FeLV disponibles en la base de datos del NCBI (National Center for Biotechnology Information). No se detectaron coincidencias entre los cebadores de FeLV diseñados y otros patógenos que pudieran interferir con una detección molecular específica. Como control positivo, se realizó el clonaje de una muestra previamente conocida como positiva para FeLV utilizando un vector pGem-T Easy® (Promega, EE.UU.) según las recomendaciones del fabricante. Los clones recombinantes fueron secuenciados utilizando la técnica de Sanger por Macrogen Inc. (Madrid, España) y, después de una búsqueda exhaustiva en BLAST® (Basic Local Alignment Search Tool), fueron utilizadas como control positivo para todas las pruebas de PCR. Durante la etapa de validación de la PCR, los cebadores fueron testados en muestras de gatos sanos como control negativo, amablemente donados por el Grupo de Investigación de Zoonosis de Tenerife (IUETSPC, ULL), para investigar su rendimiento y especificidad. Asimismo, durante este período de validación, las muestras fueron testadas simultáneamente en un laboratorio externo (Laboratorio IDEXX, Barcelona, España) para comparar los resultados obtenidos, actuando como control imparcial. Todos los resultados fueron congruentes.
PCR, secuenciación y análisis de datos
El gen env del provirus de ADN de FeLV fue amplificado utilizando el kit Fast HiFi DNA polymerase 2x Master Mix® (VWR, España), siguiendo las instrucciones del fabricante. Las condiciones de amplificación fueron: 94 °C durante 5 minutos, seguido de 35 ciclos de 94 °C durante 30 segundos, 64 °C durante 30 segundos y 72 °C durante 60 segundos, completado con una etapa de extensión final a 72 °C durante 5 minutos. Para el control negativo se utilizó agua libre de RNasa/DNasa en lugar de ADN molde. Los amplicones fueron posteriormente separados en geles de agarosa al 1.5 %, a los cuales se le añadieron 2 µl de solución de tinción de ácidos nucleicos RealSafe 20000X® (Real) por cada 100 ml de gel antes de realizar la electroforesis. Los amplicones del tamaño esperado (217 pb) fueron enviados para realizar una secuenciación Sanger a Macrogen Inc. (Madrid, España). Las secuencias obtenidas fueron comprobadas y editadas utilizando el software Chromas (Technelysium Pty Ltd) y evaluadas para determinar su similitud utilizando la herramienta BLAST®.
Construcción y análisis del árbol filogenético
Se seleccionaron regiones parciales del gen env de diferentes subgrupos enFeLV y exFeLV de la base de datos del NCBI como representativas, las cuales se utilizaron para realizar el análisis filogenético junto con las secuencias obtenidas en este estudio25. Después, se utilizó la herramienta MAFFT del software en línea ngPhylogeny.fr para limpieza y alineación múltiple de secuencias26,27. Se seleccionó como modelo óptimo de sustitución el modelo de dos parámetros de Kimura, utilizando MEGA X (Molecular Evolutionary Genetics Analysis) 28. Se eligió un retrovirus murino como grupo externo (outgroup) para enraizar el árbol filogenético22,29. Las relaciones filogenéticas fueron estimadas utilizando métodos basados en distancia (neighbor-joining algorithm) y máxima verosimilitud (maximum likelihood estimation, MLE) basado en 1000 réplicas de bootstrap28,29.
Resultados
Prevalencia y datos descriptivos
De las 96 muestras analizadas, 29 amplificaron con la PCR realizada, mostrando el tamaño esperado de 217 pb (Figura 1), sin detectarse secuencias de enFeLV. Tras el análisis PCR, se determinó que el 30.2 % de los gatos estaban infectados con FeLV.
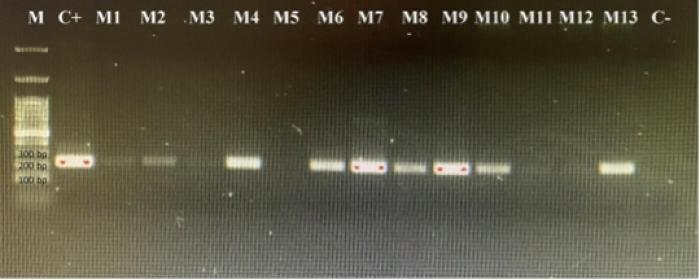
Figura 1. Visualización del fragmento amplificado por PCR de 217 pb del gen env del virus de la Leucemia Felina. M: marcador; M1 a M13: muestras positivas; M5: muestra negativa. C+: control positivo, C-: control negativo.
En el marco temporal del estudio, los años 2019 y 2021 destacaron por ser los años con el mayor número de gatos evaluados, sumando un total de 30 y 32, respectivamente. Es notable que en 2019 se registraron el doble de muestras positivas (12) en comparación con 2021 (5). La región metropolitana de Tenerife muestra un contraste significativo en la cantidad de animales evaluados en comparación con las zonas norte y sur. Esta tendencia se encuentra alineada con la ubicación de clínicas veterinarias principalmente en las zonas metropolitanas.
Un igual número de gatos machos y hembras fue sometido a la PCR, sin variaciones discernibles en los resultados según el género. El análisis de las razas reveló que los gatos Común europeo de pelo corto comprendieron el 74 % de las muestras, representando el 65.5 % de los resultados positivos. Solo se identificaron cuatro razas adicionales: Sphynx, Maine coon, Siamés y Persa, sumando seis individuos examinados, con solo dos gatos Siamés que dieron positivo por FeLV. La clasificación de los gatos por edad se realizó de la siguiente manera: cachorro (0-6 meses), joven (7 meses-2 años), adulto (3-6 años), maduro (7-10 años), mayor (11-14 años) y geriátrico (> 15 años). La mayoría de los gatos analizados tenía entre 3 y 6 años, siendo el 33 % de las muestras positivas. El siguiente grupo más representado fue el de los gatos maduros (27 muestras), con el 19 % de las muestras positivas. La edad promedio de los gatos evaluados fue de 6.5 años y notablemente, el análisis indicó que la mayoría de los gatos positivos se encontraban dentro de un rango de edad similar. Es importante destacar que ciertas muestras carecían de información descriptiva proporcionada por las clínicas veterinarias al laboratorio, y que se encuentran categorizadas como “desconocidas” en la Tabla 1.
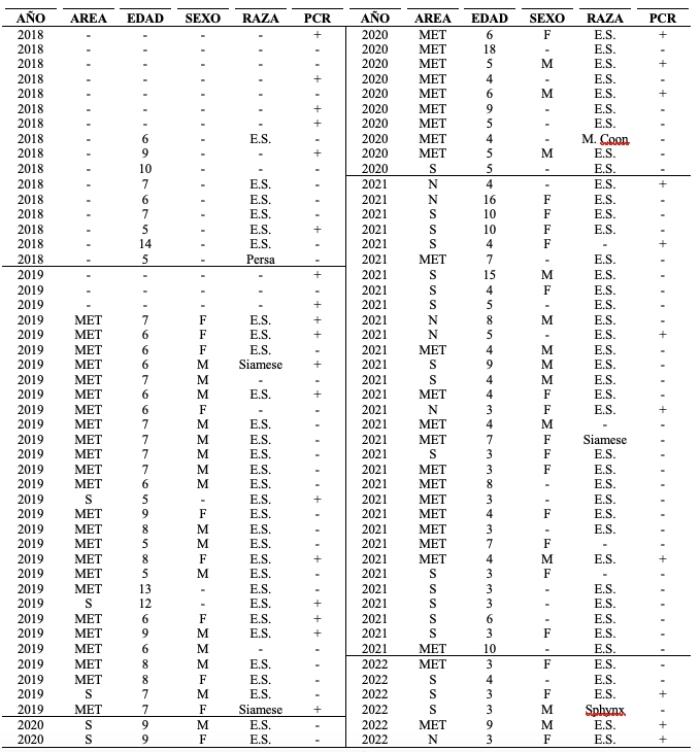
Tabla 1. Lista de muestras analizadas y los resultados obtenidos por PCR. El área considera la Isla de Tenerife dividida en tres secciones: MET = Área Metropolitana (Santa Cruz, La Laguna, El Rosario y Tegueste); N = Norte (desde Tacoronte hasta Buenavista) y S = Sur (desde Candelaria hasta Santiago del Teide). SEXO: H = Hembra y M = Macho. RAZA: E.S. = Europeo de Pelo Corto. Los guiones indican que no se proporcionó información en la solicitud de análisis para ese gato en particular.
Resultados de secuenciación
Trece muestras positivas seleccionadas al azar fueron secuenciadas y comparadas con secuencias presentes en la base de datos GenBank, mostrando una coincidencia que osciló entre 96.07 % y 98.39 %, confirmando su homología con el genoma del provirus de ADN de FeLV. Las secuencias obtenidas han sido depositadas en la base de datos bajo los números de acceso: ON799142 – ON799150 y OR493509-OR493512 (Tabla 2).
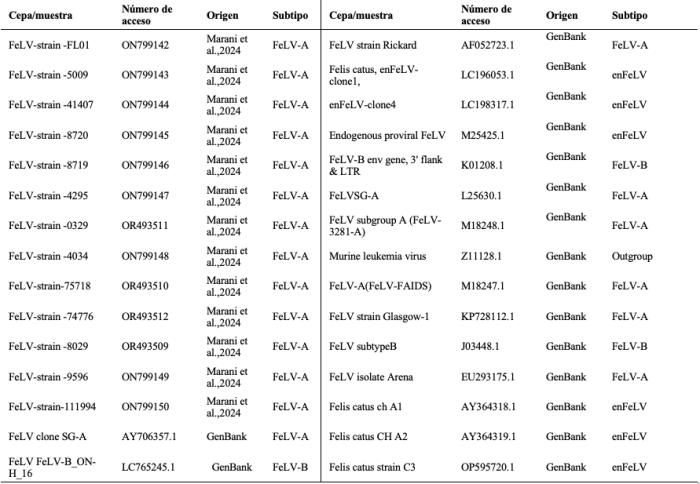
Tabla 2. Secuencias de referencia de GenBank y muestras de FeLV obtenidas en este estudio utilizadas en la construcción del árbol filogenético.
Análisis filogenético
El análisis filogenético reveló que las cepas identificadas eran altamente similares entre sí, pero lo suficientemente diferentes como para ubicarlas en dos subgrupos diferentes dentro del Subtipo-A (Figura 2). Las cepas 5009, FL-01 y 0329 de FeLV obtenidas en este estudio fueron las más cercanas a la cepa Arena (EU293175.1), mientras que FL8719 fue altamente similar a la Glasgow-1 (KP728112.1). Además, las muestras 4295, 111994 y 41407 se agruparon junto con dos muestras relacionadas con FeLV-A, la AY706357.1 y la cepa FeLV-A-AIDS (M18247.1), una variante viral relacionada con el síndrome inmunosupresor letal de FeLV23. El segundo subgrupo, que comprende las muestras 8720, 74776, 4034, 75718, 8029 y 9596 de este estudio, mostró una similitud significativa dentro de su grupo y estuvo estrechamente relacionado con el primer subgrupo de FeLV-A. Además, se observa un grupo completo más distante filogenéticamente del FeLV-A, lo que indica un mayor nivel de divergencia, agrupando la secuencia del subtipo B de FeLV y las secuencias de enFeLV de la base de datos GenBank juntas, con un soporte de valores robustos de bootstrap.
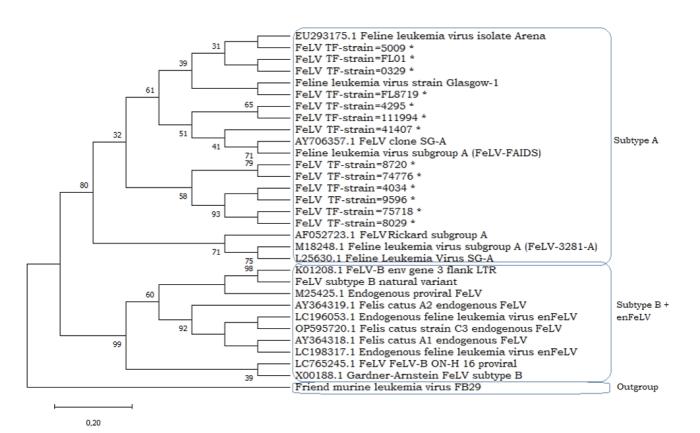
Figura 2. Análisis filogenético molecular mediante el método de máxima verosimilitud (maximum likelihood). El árbol presenta las secuencias de referencia representativas de subtipos obtenidas de la base de datos del NCBI utilizadas para realizar nuestra comparación. Las muestras de Tenerife obtenidas en este estudio están indicadas con un asterisco. Los valores de bootstrap inferiores a 30 no se muestran.
Discusión
La detección del provirus de FeLV mediante técnicas moleculares ha sido recomendada como el método estándar debido a su excelente sensibilidad y especificidad30. Sin embargo, requiere conocimientos sobre la complejidad de la enfermedad, así como habilidades para el diseño de cebadores. Según estudios previos de Arjona17, la enfermedad puede pasar desapercibida o incluso desarrollarse con síntomas inespecíficos que podrían ser difíciles de diagnosticar para los veterinarios. Estos casos pueden ocurrir en las primeras etapas de la enfermedad, ya que la viremia no se ha desarrollado lo suficiente como para ser detectada por una prueba ELISA31. Por otro lado, una vez que la infección se vuelve latente, la viremia es indetectable por cualquier prueba que no sea un ensayo de ADN-PCR proviral, ya que no depende de la carga viral. Esto es un punto importante, ya que los felinos que sufren una reactivación de la enfermedad no solo se vuelven contagiosos, sino que pueden desarrollar cualquiera de los síndromes asociados. Además, las pruebas de FeLV en cachorros siempre deben ser confirmadas por PCR, ya que pueden llevar anticuerpos de su madre en su propio torrente sanguíneo hasta 6 meses después del nacimiento2. Por lo tanto, dado que la infección por FeLV cursa con etapas de viremia variable circulante, elegimos una PCR de provirus de ADN en lugar de un ensayo de ARN. Esta elección simplifica los procedimientos de laboratorio al tiempo que garantiza una detección precisa. En relación con el diseño de cebadores, se seleccionó la región del gen env, ya que el provirus exógeno de FeLV comparte secuencias conservadas en este gen25, una característica útil empleada en análisis filogenéticos para realizar la clasificación de las cepas en subgrupos de FeLV22.
En Europa, se han descrito estudios previos sobre la prevalencia de FeLV8,9,16,31; sin embargo, las Islas Canarias no fueron incluidas y no hay datos publicados para Tenerife. En este estudio se obtuvo un porcentaje significativamente más alto de prevalencia en comparación con estudios anteriores realizados en España y otros países europeos como Alemania o Portugal30,32,33. En esos trabajos, la prevalencia de gatos positivos a FeLV variaba entre el 2.3 % y el 15.3 %. Además, en el informe paneuropeo realizado por Studer16 se informó que la prevalencia promedio observada en los países del sur de Europa no superaba el 5.5 %, utilizando un método de muestreo basado en gatos que visitan la práctica veterinaria, de manera similar a este estudio. Por otro lado, la prevalencia más baja de FeLV se encontró entre los países del norte de Europa (0.4 %-1.7 %). Considerando estos datos, una prevalencia del 30 % sugiere que podría haber algunos puntos calientes regionales de FeLV en las Islas Canarias. Vale la pena señalar que muchos de los animales incluidos en este estudio fueron llevados a clínicas veterinarias bajo sospecha de infección por FeLV, lo cual podría justificar parcialmente la alta prevalencia de FeLV observada13. Sin embargo, dado que no se compartió información clínica sobre los gatos, se incluyeron muestras de animales sanos llevados para revisiones rutinarias. De igual manera, en este estudio no se disponía de información sobre el estado de vacunación de la población. Independientemente del estado o tipo de vacuna contra el FeLV utilizada, esta no proporciona protección completa sino una producción efectiva de anticuerpos14,34. Las vacunas disponibles comercialmente han sido probadas y exploradas en numerosos estudios34–37 utilizando protocolos de PCR. Sin embargo, se ha demostrado que no previenen la integración del ADN del provirus durante las infecciones por FeLV14,38. Por lo tanto, la búsqueda de vacunas nuevas y mejoradas debe continuar, a pesar de que las actuales tienen la capacidad de reducir significativamente el impacto de la infección en gatos no expuestos y, en casos de contagio, mejorar la calidad y la expectativa de vida1,2,14.
En Tenerife, diferentes factores de riesgo podrían contribuir a estas estadísticas debido a la interacción entre gatos domésticos con acceso al exterior y numerosas colonias de gatos callejeros que viven alrededor de la isla33. Además, se sabe que la presencia de gatos asilvestrados contribuye a la propagación de enfermedades que afectan tanto a felinos salvajes como domésticos32,33,39,40, actuando ambos grupos como reservorios32,41de patógenos como Toxoplasma gondii, especies de Bartonella y otros patógenos zoonóticos33. Las temperaturas más cálidas y la disponibilidad de alimento pueden contribuir a mantener a estos animales y, por lo tanto, las infecciones y contagios, aumentando las posibilidades de propagación del virus, ya que no hay control sanitario sobre estas colonias. Esta situación es frecuente en varios países del Mediterráneo32,40, poniendo en riesgo no solo a las mascotas, sino también a los felinos en peligro de extinción que tienen una alta tasa de mortalidad frente a esta infección.
En los últimos 40 años, los ensayos de interferencia viral han sido el único método utilizado para definir y caracterizar los subgrupos de FeLV. Con el avance tecnológico de la secuenciación, las técnicas genéticas se han vuelto más confiables, rápidas, económicas y precisas que la caracterización fenotípica. Sin embargo, a pesar de estas ventajas, hasta ahora pocos estudios han investigado la reconstrucción filogenética del virus de la Leucemia Felina22. Aquí, se utilizó esta metodología para clasificar los subgrupos de FeLV basados en el enfoque de máxima verosimilitud por primera vez con cepas de Tenerife obtenidas de gatos domésticos. El árbol filogenético resultante confirmó la divergencia entre el subgrupo FeLV-A y las secuencias del enFeLV19.. Las secuencias obtenidas pertenecientes al subtipo A resultaron estar altamente conservadas, con poca variabilidad incluso entre muestras, y se observó la ausencia de secuencias recombinantes. Las secuencias pertenecientes al subtipo FeLV-B, junto con la secuencia C3 enFeLV de GenBank, se agruparon con sus ancestros, como se esperaba para las cepas recombinantes, ya que las regiones endógenas se consideran parte de elementos genómicos antiguos del gato.
Debe destacarse que las 13 secuencias incluidas en el análisis son sólo una pequeña representación de las muestras de FeLV presentes en Tenerife. Los estudios futuros deben incluir un mayor número de muestras y secuencias para mejorar la falta de información y obtener datos de prevalencia más precisos en esta región de España. Sería importante incluir, además, gatos callejeros y asilvestrados en la población a ser evaluada, ya que estas colonias son abundantes en toda la isla. La infección por FeLV tiene un impacto significativo en la población felina mundial, sin embargo, la investigación insuficiente a nivel nacional ha obstaculizado una comprensión exhaustiva de los aspectos epidemiológicos virales y la dinámica molecular en Tenerife. Este estudio representa el primer análisis filogenético de secuencias obtenidas de gatos domésticos con FeLV en las Islas Canarias, que están estrechamente relacionadas con cepas de alta virulencia y patogenicidad y presentan una gran similitud con las identificadas en el lince ibérico de Doñana, España39. Esta contribución marca un útil primer paso, dado que hay información molecular limitada sobre las secuencias de FeLV de Tenerife, principalmente debido a que la mayoría de los estudios se basan en encuestas serológicas18.
La infección por FeLV sigue siendo una enfermedad compleja de prevenir, diagnosticar y tratar. El protocolo de PCR descrito en este estudio puede servir como una herramienta valiosa para la detección en laboratorio, no solo para mascotas sino también para la investigación en felinos silvestres. Los hallazgos actuales ofrecen información valiosa y, a pesar de un tamaño muestral pequeño, se ha realizado un análisis filogenético por primera vez en el archipiélago de las Islas Canarias. Es importante reconocer la diversidad dentro de los retrovirus felinos e identificar los tipos virales prevalentes dentro de regiones específicas. Estos estudios pueden proporcionar perspectivas valiosas sobre la efectividad de los métodos diagnósticos.
Bibliografía
1. Hartmann, K. Clinical aspects of feline immunodeficiency and feline leukemia virus infection. Vet Immunol Immunopathol 143, 190–201 (2011).
2. Hartmann, K. Clinical aspects of feline retroviruses: A review. Viruses 4, 2684–2710 (2012).
3. Luaces, I. et al. Detection of Feline Leukemia Virus in the Endangered Iberian Lynx (Lynx Pardinus). J Vet Diagn Invest vol. 20 http://www.ebi.ac.uk/ (2008).
4. Lacharoje, S., Techangamsuwan, S. & Chaichanawongsaroj, N. Rapid characterization of feline leukemia virus infective stages by a novel nested recombinase polymerase amplification (RPA) and reverse transcriptase-RPA. Sci Rep 11, 1–10 (2021).
5. Erbeck, K. et al. Feline Leukemia Virus (FeLV) Endogenous and Exogenous Recombination Events Result in Multiple FeLV-B Subtypes during Natural Infection. J Virol 95, 1–10 (2021).
6. Coelho, F. M. et al. Naturally occurring feline leukemia virus subgroup A and B infections in urban domestic cats. Journal of General Virology 89, 2799–2805 (2008).
7. Chiu, E. S. Role of endogenous retrovirus in control of feline leukemia virus infection and implications for cross species transmission. Academia.Edu 1–189 (2019).
8. Roca, A. L., Pecon-Slattery, J. & O’brien, S. J. Genomically Intact Endogenous Feline Leukemia Viruses of Recent Origin. J Virol 78, 4370–4375 (2004).
9. Fuentes, I., Rubio, J. M., Ramírez, C. & Alvar, J. Genotypic characterization of Toxoplasma gondii strains associated with human toxoplasmosis in spain: Direct analysis from clinical samples. J Clin Microbiol 39, 1566–1570 (2001).
10. Chiu, E. S., Hoover, E. A. & Vandewoude, S. A retrospective examination of feline leukemia subgroup characterization: Viral interference assays to deep sequencing. Viruses 10, (2018).
11. Sohl, B. Analysis of variable regions in feline leukemia virus envelope Analysis of variable regions in feline leukemia virus envelope sequences sequences. (2017).
12. Hartmann, Katrin, Hoffmann-Lehmann, Regina. Diagnosing different courses of felv infection. (2020).
13. Miyazawa, T. & Jarrett, O. Feline leukaemia virus proviral DNA detected by polymerase chain reaction in antigenaemic but non-viraemic ('discordant’) cats. Arch Virol 142, 323–332 (1997).
14. Hofmann-Lehmann, R. et al. How molecular methods change our views of FeLV infection and vaccination. Veterinary Immunology and Immunopathology vol. 123 119–123 Preprint at https://doi.org/10.1016/j.vetimm.2008.01.017 (2008).
15. Hartmann, K. & Hoffmann-Lehmann, R. What’s New in Feline Leukemia Virus Infection. Veterinary Clinics of North America: Small Animal Practice 50, 1013–1036 (2020).
16. Studer, N. et al. Pan-European Study on the Prevalence of the Feline Leukaemia Virus Infection-Reported by the European Advisory Board on Cat Diseases (ABCD Europe). Viruses 11, 993 (2019).
17. Arjona, A. et al. Seroepidemiological survey of infection by feline leukemia virus and immunodeficiency virus in Madrid and correlation with some clinical aspects. J Clin Microbiol 38, 3448–3449 (2000).
18. Ravicini, S. et al. Prevalence of selected infectious disease agents in stray cats in Catalonia, Spain. Journal of Feline Medicine and Surgery Open Reports 2, 205511691663410 (2016).
19. Biezus, G. et al. Phylogenetic identification of feline leukemia virus A and B in cats with progressive infection developing into lymphoma and leukemia. Virus Res 329, 199093 (2023).
20. Bande, F., Arshad, S. S., Hassan, L. & Zakaria, Z. Molecular detection, phylogenetic analysis, and identification of transcription motifs in feline leukemia virus from naturally infected cats in Malaysia. Vet Med Int 2014, (2014).
21. Watanabe, S. et al. Phylogenetic and Structural Diversity in the Feline Leukemia Virus Env Gene. PLoS One 8, 1–16 (2013).
22. Cano-Ortiz, L., Tochetto, C., Roehe, P. M., Franco, A. C. & Junqueira, D. M. Could Phylogenetic Analysis Be Used for Feline Leukemia Virus (FeLV) Classification? Viruses 14, (2022).
23. Little, S. et al. 2020 AAFP Feline Retrovirus Testing and Management Guidelines. J Feline Med Surg 22, 5–30 (2020).
24. Dereeper, A. et al. Phylogeny.fr: robust phylogenetic analysis for the non-specialist. Nucleic Acids Res 36, 465–469 (2008).
25. Donahue, P. R. et al. Strong sequence conservation among horizontally transmissible, minimally pathogenic feline leukemia viruses. J Virol 62, 722–731 (1988).
26. Lemoine, F. et al. NGPhylogeny.fr: New generation phylogenetic services for non-specialists. Nucleic Acids Res 47, W260–W265 (2019).
27. Kumar, S., Stecher, G., Li, M., Knyaz, C. & Tamura, K. MEGA X: Molecular evolutionary genetics analysis across computing platforms. Mol Biol Evol 35, 1547–1549 (2018).
28. Rosenberg, M. S. Evolutionary distance estimation and fidelity of pair wise sequence alignment. BMC Bioinformatics 6, 1–9 (2005).
29. Kimura, M. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences. J Mol Evol 111–120 (1980).
30. Arjona, A. et al. Evaluation of a novel nested PCR for the routine diagnosis of feline leukemia virus (FeLV) and feline immunodeficiency virus (FIV). J Feline Med Surg 9, 14–22 (2007).
31. Arjona, A. et al. Seroepidemiological Survey of Infection by Feline Leukemia Virus and Immunodeficiency Virus in Madrid and Correlation with Some Clinical Aspects. JOURNAL OF CLINICAL MICROBIOLOGY https://journals.asm.org/journal/jcm (2000).
32. Millán, J. & Rodríguez, A. A serological survey of common feline pathogens in free-living European wildcats (Felis silvestris) in central Spain. Eur J Wildl Res 55, 285–291 (2009).
33. Tobajas, J. Riesgos genéticos y sanitarios asociados al gato asilvestrado (Felis silvestris catus): el caso de los felinos salvajes de la península ibérica. Chronica naturae 82, 63–82 (2016).
34. Torres, A. N., O’Halloran, K. P., Larson, L. J., Schultz, R. D. & Hoover, E. A. Feline leukemia virus immunity induced by whole inactivated virus vaccination. Vet Immunol Immunopathol 134, 122–131 (2010).
35. Hofmann-Lehmann, R. et al. Reassessment of feline leukaemia virus (FeLV) vaccines with novel sensitive molecular assays. Vaccine 24, 1087–1094 (2006).
36. Hofmann-Lehmann, R. et al. Vaccination against the feline leukaemia virus: Outcome and response categories and long-term follow-up. Vaccine25, 5531–5539 (2007).
37. Sparkes, A. H. Feline leukaemia virus and vaccination. in Journal of Feline Medicine and Surgery vol. 5 97–100 (W.B. Saunders Ltd, 2003).
38. Acevedo-Jiménez, G. E., Sarmiento-Silva, R. E., Alonso-Morales, R. A., Córdova-Ponce, R. & Ramírez-Álvarez, H. Detection and genetic characterization of feline retroviruses in domestic cats with different clinical signs and hematological alterations. Arch Virol 168, (2023).
39. Meli, M. L. et al. Feline leukemia virus and other pathogens as important threats to the survival of the critically endangered Iberian lynx (Lynx pardinus). PLoS One 4, (2009).
40. Duarte, A., Fernandes, M., Santos, N. & Tavares, L. Virological Survey in free-ranging wildcats (Felis silvestris) and feral domestic cats in Portugal. Vet Microbiol 158, 400–404 (2012).
41. Palmer, M. V., Stoffregen, W. C., Carpenter, J. G. & Stabel, J. R. Isolation of Mycobacterium avium subsp paratuberculosis (Map) from feral cats on a dairy farm with Map-infected cattle. J Wildl Dis 41, 629–635 (2005).
 by Multimédica
by Multimédica
