Sanidad animal y salud pública: el SARS-CoV-2 en los animales
Resumen breve
Las zoonosis procedentes de los pequeños animales pueden surgir de la forma más insospechada y acarrear graves consecuencias. El nuevo estilo de vida y la interdependencia entre personas y animales de compañía, así como los numerosos factores que condicionan esa relación, se han combinado para crear un terreno propicio a la aparición de patógenos zoonósicos.Índice de contenidos
¿Cómo empezo todo? Introducción.
Las zoonosis procedentes de los pequeños animales pueden surgir de la forma más insospechada y acarrear graves consecuencias. El nuevo estilo de vida y la interdependencia entre personas y animales de compañía, así como los numerosos factores que condicionan esa relación, se han combinado para crear un terreno propicio a la aparición de patógenos zoonósicos.
En este sentido, y debido a la importancia de la crisis sanitaria que nos afecta vamos a centrar nuestra atención en una zoonosis emergente: el estudio de la infección por SARS-CoV-2 y su relación con los animales de compañía.
Un dato científico previo muy esclarecedor: en 2007, un artículo científico publicado en la revista «Clinical Microbiology Review» por Vincent et al. de la Universidad de Hong-Kong, advertía del riesgo potencial de recombinación de los coronavirus para generar nuevos genotipos y brotes. La presencia de un reservorio de SARS-CoV en murciélagos herradura unido a la ancestral cultura gastronómica de comer mamíferos en el sur de China, suponía una auténtica bomba. Finalmente, los autores concluían que la posibilidad de reemergencia de SARS y otros nuevos virus procedentes de animales o laboratorios requería un alto grado de alerta.

Lógicamente, y así lo han comunicado las autoridades sanitarias a través de la evidencia científica, parece poco probable que el SARS-CoV-2 haya surgido de un laboratorio. La Sociedad Española de Virología nos argumenta este hecho (Figura 1).
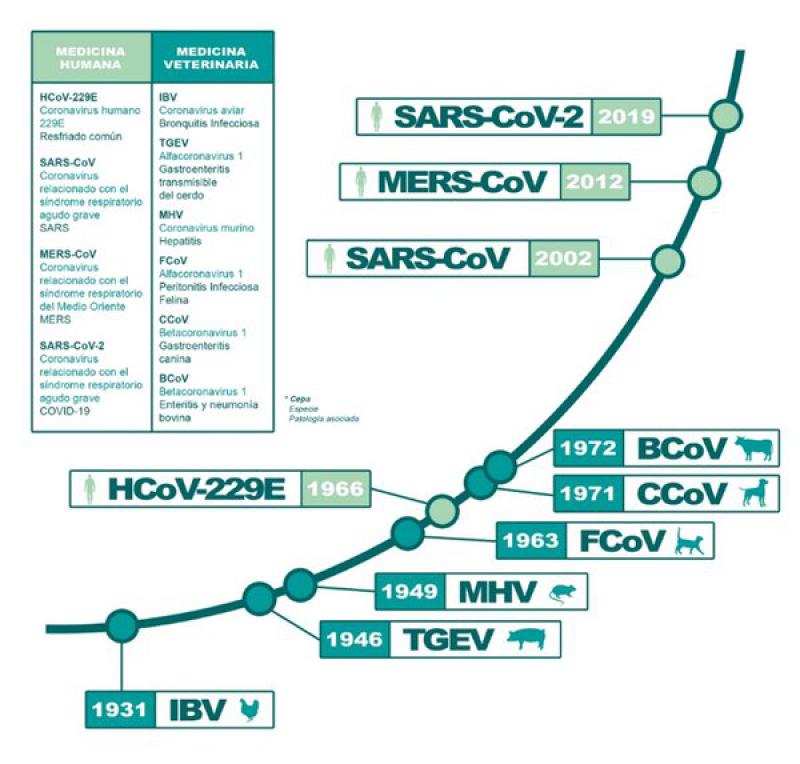
Son varios los coronavirus que han causado y siguen causando serios problemas en humanos y animales en las últimas dos décadas. Los ejemplos más conocidos son el coronavirus del Síndrome Respiratorio Agudo Severo (SARS-CoV), el coronavirus del Síndrome Respiratorio de Oriente Medio (MERS-CoV), el virus de la Diarrea Epidémica Porcina (PEDV) y actualmente la COVID-19 (SARS-CoV-2).
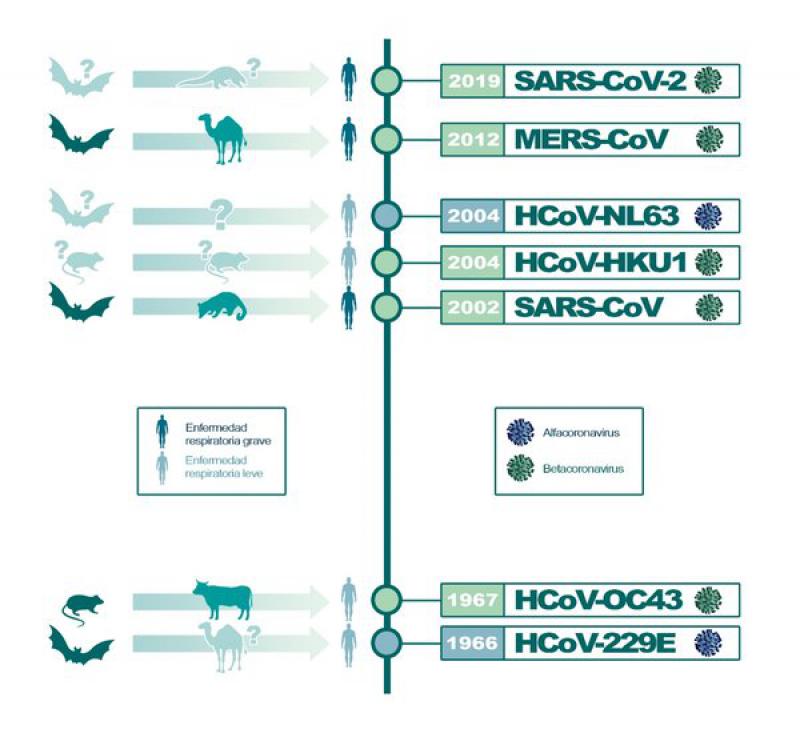
La epidemia del SARS-CoV de 2003 aumentó el interés en la búsqueda de nuevos coronavirus; interés que de nuevo descendió a nivel cero cuando esta epidemia se extinguió tan rápido como empezó. Antes de esta epidemia del SARS-CoV solo se conocían dos coronavirus que infectaban a los humanos: el coronavirus humano HCoV-229E y el HCoV-OC43.
Posteriormente a estos se descubrieron otros dos coronavirus humanos, el HCoV-NL63 y HCoVHKU1. Estos cuatro virus son responsables de alrededor de un 20 % de los casos de infecciones respiratorias de vías altas que nos afectan. Aunque en la mayoría de los casos producen resfriados comunes, en algunas ocasiones pueden generar cuadros respiratorios más graves como bronquiolitis y neumonías. Es más que probable que estos virus hayan estado circulando en humanos durante mucho tiempo antes de su descubrimiento.
El 23 de septiembre de 2012, la Organización Mundial de la Salud (OMS) anunció el descubrimiento de un nuevo coronavirus, el MERS-CoV, en dos pacientes que murieron a causa de una enfermedad misteriosa y rápidamente mortal en Oriente Medio. El primero de ellos fue un paciente saudí de 60 años que falleció tras padecer una neumonía con fallo renal agudo. El coronavirus del MERS-CoV presenta una tasa de letalidad aún mayor (> 35 %) que el SARS (9.6 %), lo que puede explicarse en parte por la alta prevalencia de comorbilidades médicas entre los pacientes infectados.
Afortunadamente el MERS-CoV no se ha transmitido de persona a persona con tanta eficacia como lo hizo el SARS-CoV o lo hace ahora el SARS-CoV-2. Las investigaciones llevadas a cabo en aquel momento de su primera descripción revelaron que el virus estaba más estrechamente relacionado con los betacoronavirus del linaje C, el coronavirus de murciélago Tylonycteris HKU4 (Tylonycteris pachypus y Tylonycteris robustula) y el coronavirus del murciélago Pipistrellus HKU5 (Pipistrellus abramus), previamente identificados en Hong Kong. Posteriormente, los dromedarios (Camelus dromedarius) fueron identificados como la fuente de algunos casos humanos.
En diciembre de 2019 surge el SARS-Coronavirus-2 (SARS-CoV-2) en China, causando una oleada de muertes en Wuhan y de forma menos severa en otras regiones adyacentes. A partir de ese momento este coronavirus se disemina por todo el mundo y el 11 de marzo de 2020 la OMS declara el estado de pandemia, paralizando todo el planeta. En la fecha que se escribe este artículo, el SARS-CoV-2 ha producido más de 172 millones de casos con más de 3.7 millones de muertes. Aun así y a pesar de estos datos, tenemos que estar «contentos» de que este virus no presente la tasa de letalidad de sus «parientes»: el MERS-CoV o el propio SARS-CoV de 2003.
Se piensa que el SARS-CoV-2 emergió de una fuente de origen animal y posteriormente saltó a la población humana. Pese a ser genéticamente cercano a los virus que se han aislado de los murciélagos del género Rhinolophus, aún no se han establecido de manera concluyente los orígenes animales del SARS-CoV-2 ni su relación epidemiológica con la población humana.
La pandemia actual de la COVID-19 se mantiene a través de la transmisión específica humano a humano. Sin embargo, varios países han notificado casos de infecciones animales por SARS-CoV-2. De hecho, se ha demostrado la susceptibilidad de ciertas especies animales a la infección por SARS-CoV-2, tanto de forma natural como por infección experimental. En este sentido, estudios experimentales han demostrado que importantes especies de producción (aves de corral y cerdos, entre otras) no son susceptibles a la infección por SARS-CoV-2; aunque son necesarios más estudios para comprender la implicación del SARS-CoV-2 en diferentes especies animales.
Por todo ello, es esencial la vigilancia y el seguimiento epidemiológico de las infecciones en animales con el fin de conocer mejor su importancia en el marco del binomio «sanidad animal y salud pública». La evidencia derivada de los estudios de evaluación del riesgo a partir de investigaciones epidemiológicas y estudios experimentales indican que los animales no cumplen un papel significativo en la propagación de SARS-CoV-2, que se mantiene por transmisión de humano a humano (OIE, mayo 2021).
La infección por SARS-CoV-2 no está incluida en la lista de enfermedades de la Organización Mundial de la Sanidad Animal (OIE). Sin embargo, conforme a las obligaciones de notificación de los Miembros que figuran en los artículos 1.1.4. y 1.1.6. del Código Sanitario para los Animales Terrestres relacionados con las enfermedades emergentes, la enfermedad debe notificarse a la OIE a través del Sistema Mundial de Información Sanitaria o por correo electrónico.
En este artículo pretendemos abordar la infección/enfermedad (COVID-19) y otros coronavirus en los animales a través de una breve reseña, destacando sus aspectos epidemiológicos y de control, así como desglosando algunas de las investigaciones actualizadas sobre el tema basadas en infecciones naturales y experimentales publicadas en revistas especializadas de índole científico.
¿Pero qué son los coronavirus? Una breve mirada a la virología
Los Coronavirus (CoV) son un grupo diverso de patógenos capaces de infectar a muchas especies de mamíferos y aves. Causan una variedad de enfermedades, produciendo desde cuadros respiratorios, digestivos y/o renales, a otros menos frecuentes como neurológicos y cardiovasculares.
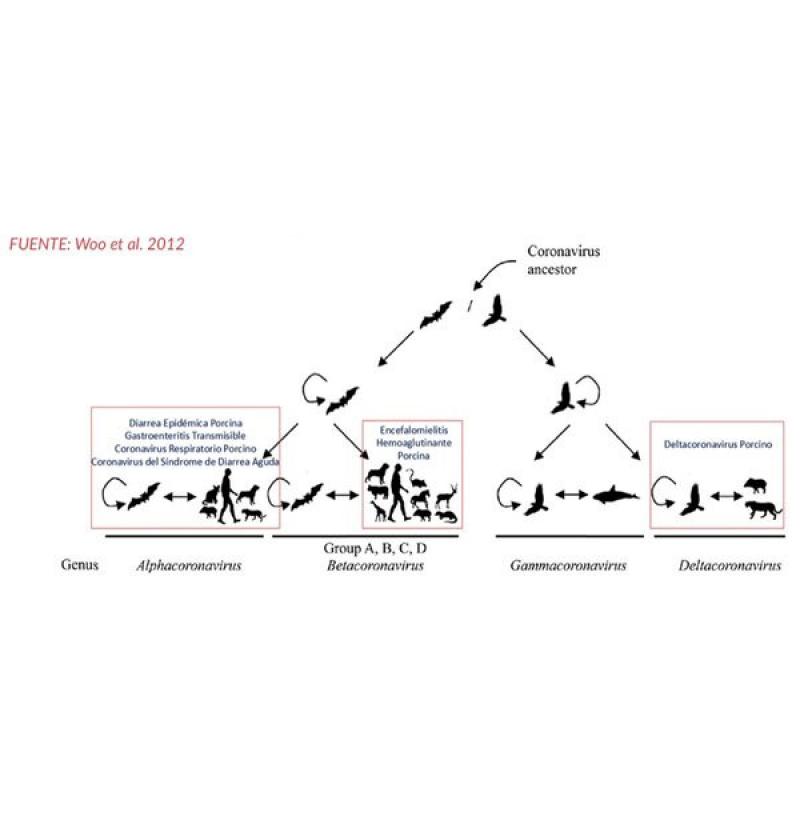
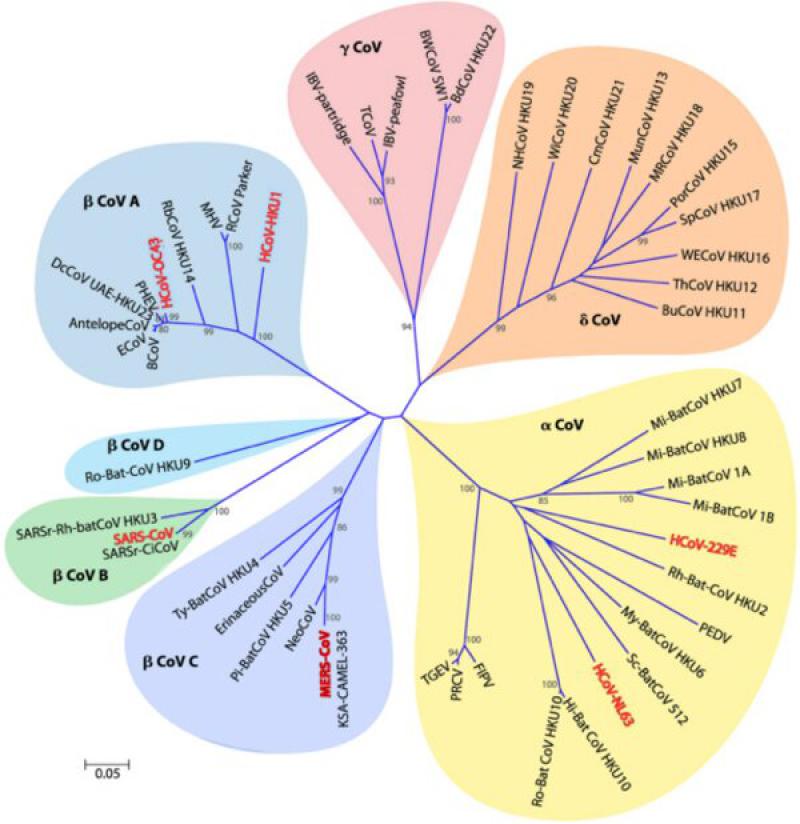
Desde el punto de vista taxonómico, los CoV pertenecen al orden Nidovirales, familia Coronaviridae. Dentro de esta familia se organizan dos subfamilias: Orthocoronavirinae y Letovirinae. La subfamilia Orthocoronavirinae agrupa cuatro géneros: Alpha-, Beta-, Gamma- y Deltacoronavirus, cada uno de los cuales a su vez incluye a distintas especies virales.
En general, se acepta que los alpha- y betacoronavirus solo infectan especies de mamíferos, mientras que los gamma- y deltacoronavirus infectan aves, aunque algunos pertenecientes a estos dos últimos géneros también pueden infectar a algunas especies de mamíferos (por ejemplo, el cerdo y la ballena beluga) (Figura 2-3, Tabla 1).
| Tabla 1. Clasificación de coronavirus y especies (virus) que afectan.. |
| Género | Especie | Hospedador |
| Alphacoronavirus | Virus de la gastroenteritis transmisible porcina (TGEV) Virus de la diarrea epidémica porcina (PEDV) Coronavirus felino (FCoV) Coronavirus canino (CCoV) Coronavirus humano 229E (HCoV-229E) | Cerdo Cerdo Gato Perro Humano |
| Betacoronavirus | Coronavirus bovino (BCoV) Virus de la encefalomielitis hemalglutinante porcina (HEV) Coronavirus equino (ECoV) Virus de la hepatitis murina (MHV) Coronavirus de rata (RtCoV) Coronavirus humano OC43 (HCoV-OC43) | Vaca Cerdo Caballo Roedores Roedores Humano |
| Gammacoronavirus | Coronavirus Wigeon HKU20 Coronavirus de Bulbul HKU11 (BuCoV HKU11) Coronavirus de garza nocturna HKU19 Coronavirus Munia HKU13 (MunCoV HKU13) | Aves Aves Aves Aves |
| Deltacoronavirus | Coronavirus aviar Coronairus de pato 2714 Coronavirus de ganso CB17 Coronavirus de la ballena Beluga SW1 | Aves Aves Aves Ballena Beluga |
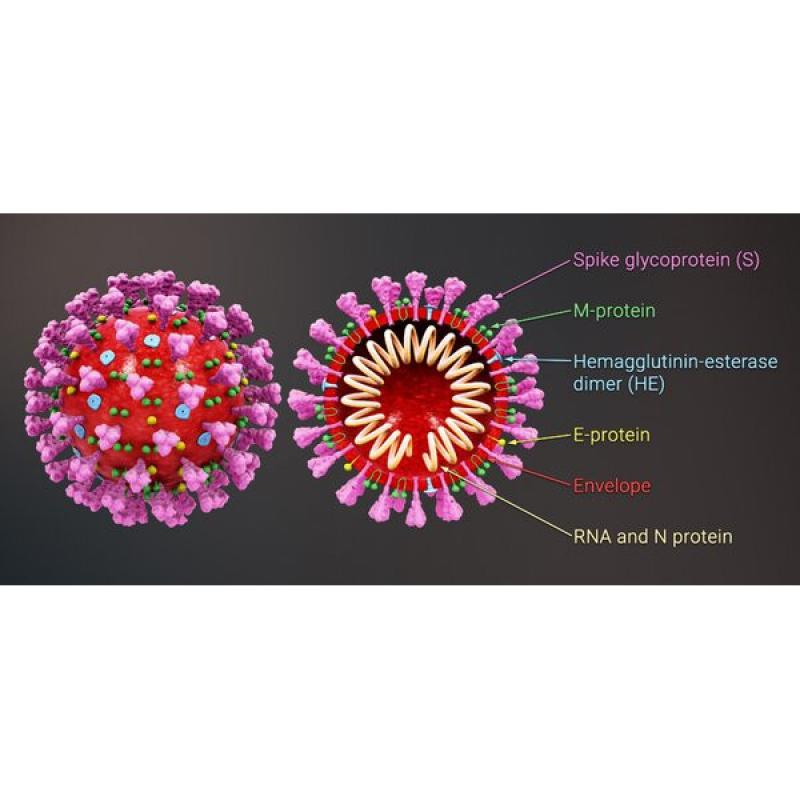
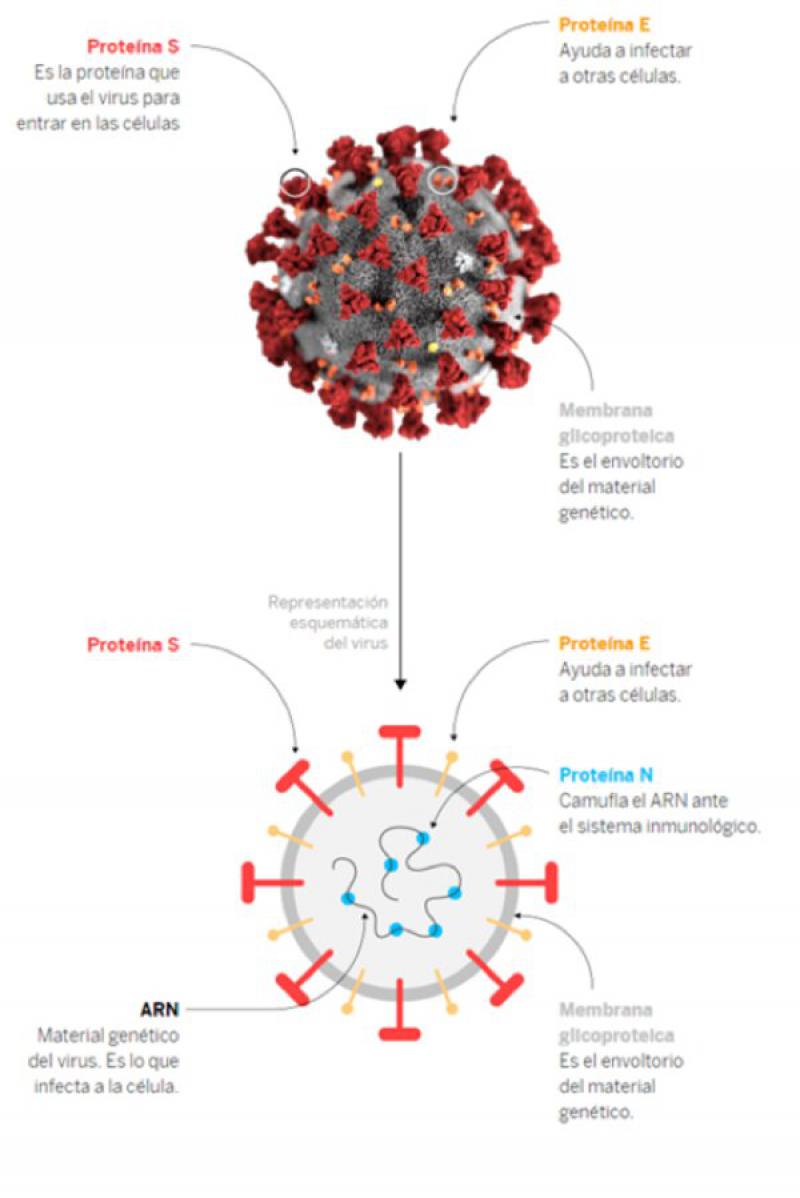
Son virus grandes, envueltos, de sentido positivo, y de cadena sencilla de ARN, moderadamente pleomórficos y con cierta tendencia a la esfericidad. Presenta unas proyecciones en forma de garrote (peplómeros), formados por la proteína trimérica S (Spike o espiga). Su nucleocápside es simétrica y helicoidal y está formada por ARNs (de cadena simple o monocatenario) y sentido positivo (Figura 4).
Resistencia a agentes químicos
Inactivación del SARS-CoV-2:
- Etanol (60-70 %); peróxido de hidrógeno (0.5 %); hipoclorito de sodio (0.1 %).
- Cloruro de benzalconio (0.05-0.2 %; digluconato de clorhexidina (0.02 %).
Supervivencia
En condiciones experimentales, el SARS-CoV-2 permanece viable en el ambiente tras aerosolización durante al menos 180 minutos. La experiencia con otros CoV tales como SARS-CoV, MERS-CoV, o coronavirus humanos endémicos muestran que:
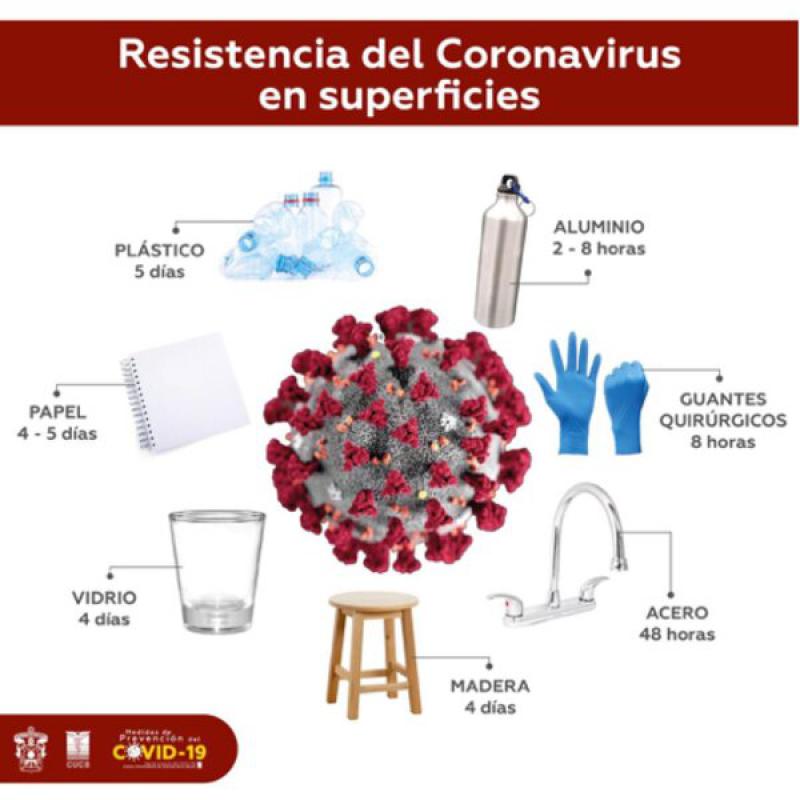
- Pueden persistir en superficies tales como el metal, el vidrio o el plástico hasta 9 días, pero pueden inactivarse eficazmente a través de procedimientos de desinfección de superficies, como los mencionados anteriormente (Figura 5).
- Los SARS-CoV permanecen infectivos durante 14 días a 4 °C, pero solo 2 días a 20 °C en aguas residuales.
Epimediología
Origen animal de los coronavirus humanos (HCoV)
Pese a que la evidencia disponible actualmente sugiere que el SARS-CoV-2 emergió de una fuente animal, está todavía no se ha identificado. Todos los indicios científicos señalan al murciélago como reservorio primario, al igual que ha ocurrido en el caso del SARS-CoV-1 y el MERS.
La pandemia actual se mantiene a través de la transmisión de SARS-CoV-2 de persona a persona por medio gotitas y secreciones respiratorias que se liberan al toser, estornudar y hablar. Los datos de secuencia genética muestran que el SARS-CoV-2 es un pariente cercano de otros coronavirus que circulan en poblaciones de murciélagos del género Rhinolophus (murciélagos de herradura).
Conviene recordar que los murciélagos son los mamíferos más numerosos en el reino animal, con casi mil especies diferentes y por ello suponen un nicho ecológico idóneo en el que circulan potenciales virus emergentes con capacidad de reorganización (recombinación), al igual que ocurre en el caso de las aves silvestres y los subtipos del virus Influenza.

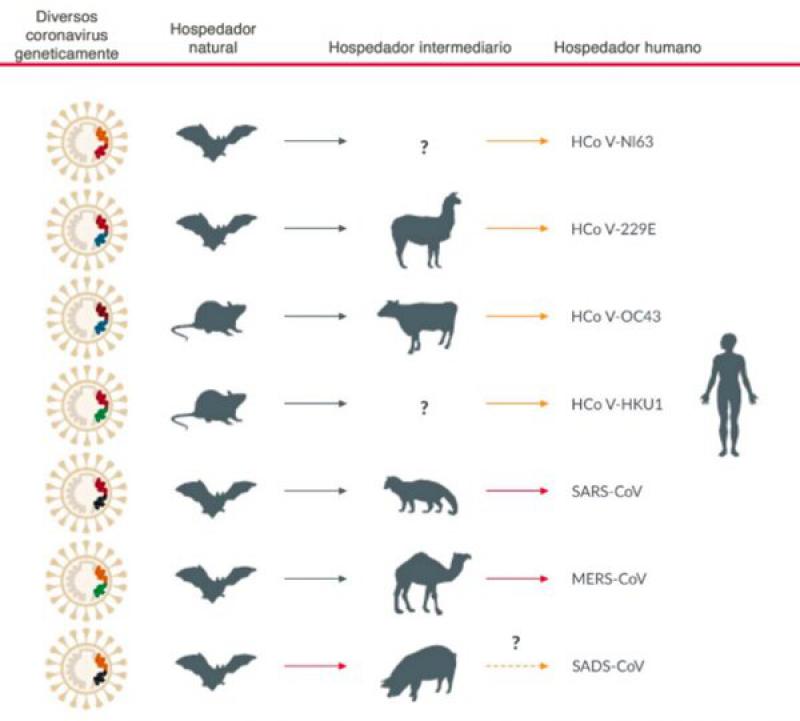
En el caso del reservorio secundario o accidental se hipotetiza que este sea el pangolín, por cierto, el mamífero más «traficado» del mundo. Las escamas del pangolín son muy demandadas en algunos países asiáticos, como China o Vietnam, donde se usan como medicina tradicional para el tratamiento de remedios como el asma, el reumatismo o la artritis. Recientemente, también se ha atribuido este carácter de reservorio secundario o accidental al perro mapache, también conocido como perro japonés; estos animales son una industria importante en China, donde son criados en granjas y capturados en la naturaleza por su pelaje (Figura 6).
Años antes del descubrimiento de la cepa B814 del coronavirus humano HCoV-229E, a partir de la secreción nasal de pacientes que habían contraído un resfriado común, ya se habían aislado diferentes coronavirus en especies como pavos, ratones, vacas, cerdos, gatos y perros.
La mayoría de los coronavirus humanos (HCoV), de los cuales se han identificado hasta la fecha siete, causan trastornos que van desde enfermedades respiratorias (resfriado común, bronquitis y bronquiolitis, neumonías), a digestivas y neurológicas, siendo las personas en estado de inmunocompromiso, ancianos y niños los mayormente afectados por formas graves. Los HCoV se reconocen como los virus de evolución más rápida.
HCoV de baja patogenicidad.
Además de los conocidos y temidos HCoV altamente patógenos (SARS-CoV, MERS-CoV y SARS-CoV-2), se han descrito otros cuatro (HCoV-229E, HCoV-OC43, HCoVNL63 y HCoV-HKU1), que presentan normalmente una baja patogenicidad, habiéndose estudiado su origen zoonósico. Así, la evidencia filogenética indica que tanto el HCoV-NL63 como el HCoV-229E podrían haberse originado a partir de los coronavirus de murciélago, mientras que los virus HCoV-OC43 y HCoV-HKU1 se han encontrado relacionado con los roedores. Se ha descrito que un coronavirus quiróptero denominado ARCoV.2 (Appalachian Ridge CoV), detectado en murciélagos tricolor de América del Norte, muestra una estrecha relación genética con el HCoV-NL63. Por otro lado, el HCoV-229E se ha relacionado genéticamente con otro coronavirus de murciélago denominado Hipposideros/Ghana Kwam/19/2008, que se detectó en Ghana, sospechándose también que los camélidos eran su hospedador intermediario (Figura 7).
Estos cuatro HCoV que causan síntomas leves se han adaptado bien a los seres humanos, aunque desde otra perspectiva, también podría ser cierto que los humanos se han adaptado bien a estos cuatro HCoV. En otras palabras, ambos podrían ser los supervivientes de antiguas pandemias de HCoV.
Para que esto suceda, los HCoV tienen que replicarse en humanos en la medida suficiente para permitir la acumulación de mutaciones adaptativas que contrarresten los factores de restricción del hospedador. En este sentido, cuanto más persista el brote de SARS-CoV-2 y a más personas infecte, mayor será la probabilidad de que se adapte completamente a los humanos. Si se adapta bien, su transmisión en humanos sería difícil de detener mediante cuarentena u otras medidas de control de la infección.
Los cuatro HCov menos letales circulan en poblaciones humanas provocando cuadros respiratorios de vías altas leves (resfriado común) en sujetos inmunocompetentes. Estos virus no necesitan un reservorio animal. Por el contrario, el SARS-CoV y el MERS-CoV altamente patógenos no se han adaptado bien a los humanos y su transmisión dentro de estos no puede sostenerse. Necesitan mantener y propagarse en sus reservorios zoonósicos y buscar la oportunidad de propagarse a objetivos humanos susceptibles, posiblemente a través de uno o más hospedadores intermediarios y amplificadores.
El SARS-CoV-2 tiene características similares tanto al SARS-CoV y MERS-CoV como a los cuatro HCoV descritos que producen infecciones adquiridas en la comunidad. Por el momento el SARS-CoV-2 es altamente transmisible, como estos HCoV adquiridos en la comunidad, y más virulento que estos, aunque afortunadamente menos virulento que el SARS-CoV y el MERS-CoV. Queda por ver si se adaptará completamente a los humanos y quedará como un virus estacional circulante sin necesidad de un reservorio u hospedador intermediario.
SARS-CoV
En relación con su origen, los estudios epidemiológicos realizados revelaron que el caso índice de SARS-CoV tenía un historial de contacto con animales de caza. Estudios posteriores de seroprevalencia indicaron que los comerciantes de animales tenían una mayor prevalencia de IgG anti-SARS-CoV en comparación con la población general.
Las civetas de palma enmascaradas (Paguma larvata) y un perro mapache en los mercados de animales vivos se identificaron por primera vez como portadores de virus similares o casi idénticos al SARS-CoV. Esto fue apoyado de forma indirecta por el hecho de que después de eliminar a todas las civetas de los mercados chinos, no se declararon más casos SARS-CoV en esas localidades.
También sabemos que las civetas de palma enmascaradas silvestres y de granjas, sin exposición previa a determinados ambientes como los mercados de animales vivos, fueron en gran medida negativas para el SARS-CoV, lo que sugiere que estos animales solo pueden servir como hospedador amplificador intermedio, pero no como hospedador natural y reservorio del SARS-CoV.
En particular, dado que el 80 % de los diferentes animales en los mercados de Guangzhou tienen anticuerpos anti-SARS-CoV, no se puede excluir la posibilidad de que múltiples especies de pequeños mamíferos también sirvan como hospedadores amplificadores intermedios de este virus.
Los estudios posteriores basados en la búsqueda del hospedador animal natural del SARS-CoV, revelaron un coronavirus quiróptero estrechamente relacionado, denominado Rhinolophus- CoV-HKU3 (SARSr-Rh-BatCoV HKU3), que existe en los murciélagos de herradura chinos. Estos murciélagos son positivos para anticuerpos anti-SARS-CoV y presentan secuencias del genoma de SARSr-Rh-BatCoV HKU3.
Este y otros coronavirus de murciélagos comparten entre el 88-92% de homología de secuencia de nucleótidos con el SARS-CoV. También se han identificado varios coronavirus similares al SARS-CoV (SL-CoV) en murciélagos, pero ninguno, excepto uno designado WIV1, puede aislarse como virus infectivo. Curiosamente, los sueros de pacientes convalecientes con SARS-CoV fueron capaces de neutralizar el WIV1. Hasta ahora, este virus WIV1 representa el ancestro más estrechamente relacionado del SARS-CoV en murciélagos, compartiendo una homología de secuencia de nucleótidos del 95 %. A pesar de la alta homología entre estos dos virus, generalmente se cree que WIV1 no es el virus parental inmediato del SARS-CoV. Estos estudios sentaron las bases para el nuevo concepto de que los murciélagos albergan patógenos humanos emergentes.
MERS-CoV
Los estudios filogenéticos agrupan a MERS-CoV en el mismo grupo que bat CoV-HKU4 y bat CoV-HKU5. Bat CoV-HKU4 y MERS-CoV utilizan el mismo receptor para la entrada del virus en las células del hospedador: la dipeptidil peptidasa 4 (DPP4). El virus MERS-CoV y el coronavirus más «cercano» genéticamente (el bat CoV-HKU25), comparten sólo un 87% de homología de secuencia de nucleótidos, lo que implica que los murciélagos podrían no ser el reservorio inmediato del MERS-CoV.
Por otra parte, estudios realizados en Oriente Medio han demostrado que los dromedarios son seropositivos para anticuerpos neutralizantes específicos de MERS-CoV, al igual que los camellos de origen Oriente Medio en varios países africanos. Además, se aislaron MERS-CoV infectivos, idénticos al virus encontrado en humanos, a partir de hisopos nasales de dromedarios, lo que indica que los camellos sirven como reservorios auténticos del MERS-CoV.
Los camellos infectados transmiten virus no solo a través de la vía respiratoria sino también a través de la vía fecal-oral. Muchos casos confirmados de MERS no tienen antecedentes de contacto con camellos antes de la aparición de los síntomas, lo que habla en favor de la transmisión de persona a persona, o a través de otras rutas desconocidas que involucran especies animales no reconocidas que albergan el MERS-CoV.
SARS-CoV-2
El SARS-CoV-2 comparte una homología de nucleótidos del 96.2 % con el bat-CoV-RaTG13 aislado de murciélagos Rhinolophus affinis. Otro coronavirus, el RmYN02, descubierto recientemente en murciélagos de la especie Rhinolophus malayanus, estudiado en la provincia de Yunnan, muestra una homología del 93.3 % con el SARS-CoV-2. Como en los casos del SARS-CoV y MERS-CoV, la divergencia de secuencia entre SARS-CoV-2 y bat-Cov-RaTG13 es demasiado grande para establecer una relación «parental».
Esto quiere decir que es posible que los murciélagos no sean los hospedadores reservorios inmediatos del SARS-CoV-2 a menos que en el futuro se encuentren coronavirus de murciélago casi idénticos. Presumiblemente, los hospedadores intermediarios del SARS-CoV-2 deberían estar entre las especies de vida silvestre que se venden y matan en el mercado mayorista de mariscos de Huanan, lugar donde se describieron los primeros casos de COVID-19, lo que indica una probable transmisión de animal a humano. Varios estudios recientes basados en la secuenciación metagenómica han sugerido, como señalábamos antes, que un grupo de pequeños mamíferos en peligro de extinción conocidos como pangolines (Manis javanica) también podrían albergar Betacoronavirus ancestrales relacionados con el SARS-CoV-2.
El coronavirus encontrado en pangolines presenta dos sublinajes llamados Guangdong y Guangxi. El primero de ellos presenta una homología del 92.4 % con el SARS-CoV-2, mientras que el segundo solo un 85.5 %. Estos coronavirus del pangolín están también estrechamente relacionados con el bat-CoV-RaTG13, mostrando un 90 % de identidad a nivel de secuencia de nucleótidos. No se puede excluir la posibilidad de que el pangolín sea uno de los hospedadores intermediarios del SARS-CoV-2, sin embargo, actualmente no hay evidencia que apoye un origen directo de este virus en esta especie animal, debido al alto grado de divergencia de secuencia entre el SARS-CoV-2 y los descritos en el pangolín.
Por si esto fuese poco, la distancia entre el SARS-CoV-2 y el bat-CoV-RaTG13 es incluso más corta que entre el SARS-CoV-2 y los mencionados coronavirus relacionados con el SARS-CoV-2 del pangolín, cosa que apoya aún más el que es poco probable el origen directo del SARS-CoV-2 en este animal. A fecha de hoy no se ha establecido la vía evolutiva del SARS-CoV-2 en murciélagos, pangolines y otras especies mamíferas. Sí que se ha descrito que estos tres virus (Betacoronavirus relacionados con el SARS-CoV-2 del pangolín, SARS-CoV-2 y bat-CoV-RaTG13), son los que presentan una mayor homología de secuencia de todo el genoma.
Hasta el momento, no se dispone de suficiente evidencia científica para identificar el origen del SARS-CoV-2 o explicar la vía de transmisión original a los humanos y que, además, podría haber implicado un huésped intermediario (pangolines, perro mapache japonés, entre otros).
Varias especies animales han dado resultados positivos al SARS-CoV-2, la mayoría como resultado de un contacto estrecho con humanos infectados por SARS-CoV-2. Además, resultados preliminares de estudios experimentales han mostrado que las aves de corral y los cerdos no son susceptibles a la infección por SARS-CoV-2. La lista de especies animales para las que se encuentra información disponible sobre la infección natural o experimental se presenta en la Tabla 2.
| Tabla 2. Infección natural y/o experimental por SARS-CoV-2 en animales. (Fuente: OIE, Ficha técnica del SARS-CoV-2). |
| Especies | Tipo de infección (experimental (laboratorio)/natural (campo)) | Susceptibilidad de la infección (ningúna/extremadamente baja/baja/media/alta) | Signos clóinicos | Transmisión |
| Visión americano (Neovision visión) | Natural y experimental | Alta | Sí, (en algunos casos) | Sí entre visiones y de visiones al homnbre |
| Hurones | Natural y experimental | Alta | Sí (sólo en algunos casos) | Sí, entre hurones |
| Perros mapache (Nyctereutes procyonoides) | Experimental | Alta | No | Sí, entre perros mapache |
| Conejos (conejo blanco de Nueva Zelanda, Oryctolagus cuniculus) | Experimental | Mediana | No | No |
| Cerdos (Cerdos mestizos Yorkshire americano, Sus scrofa) | Experimental | Extremadamente baja | No | No |
| Bovinos (bos Taurus) | Experimental | Extremadamente baja | No | No |
| Aves de corral (pollos, patos y pavos) | Experimental | Ninguna | No | No |
| Gatos (domésticos) | Natural y experimental | Alta | Sí (pero no se han observado en todos los casos) | Sí, entre gatos |
| Hámsteres sirios (Mesocricetus auratus) | Experimental | Alta | Sí (de ninguno a muy leves en algunos casos, dependiendo de la edad) | Sí, entre hámsteres |
| Perros | Natural y experimental | Baja | Sí ( pero no se han observado en todos los casos) | No |
| Grandes felinos (tigres, leones, leopardos de las nieves y pumas) | Natural | Alta | Sí, en la mayoría de los casos | Sí, entre animales |
| Murciélago egipcio de la fruta (Rousettus aegyptiacus) | Experimental | Alta | No | Sí, entre murciélagos fugitivos |
| Gorilas (Gorilla gorilla) | Natural | Alta | Sí | Sí |
| Ciervos de cola blanca (Odocoileus virginianus) | Experimental | Alta | Sí, a otros ciervos de cola blanca | |
| Rití común (Callithrix jacchus) | Experimental | No | No | No |
| Macacos (Macaca fascicularia y Macaca mulatta) | Experimental | Alta | Sí | Sí |
| Nutrias asiáticas de uñas cortas (Aonyx cinereus) | Natural | Alta | Sí | ? |
| Topillos rojos (Myodes glareolus) | Experimental | Media | No | No |
| Ratones ciervo (Peromyscus maniculatus) | Experimental | Alta | No | Sí, a otros ratones ciervos |
Ha habido informes esporádicos de animales de compañía y de animales silvestres cautivos infectados por SARS-CoV-2. Con respecto a los animales de producción, hasta la fecha, el SARS-CoV-2 sólo ha afectado granjas de visones y hurones en diferentes países europeos y en Estados Unidos (EE. UU.), con una alta morbilidad y una baja mortalidad.
Fuentes y vías de transmisión
La información sobre las vías de transmisión del SARS-CoV-2 entre animales es limitada. No obstante, al igual que para otros virus respiratorios, parece transmitirse a animales y entre animales por contacto directo a través de secreciones respiratorias (gotitas de aerosol). Se ha encontrado SARS-CoV-2 en secreciones del tracto respiratorio y en heces.
Determinantes virales de la transmisión.
La capacidad de mutación y de recombinación genética de los coronavirus ha llevado a la aparición continua de nuevas cepas con mayor virulencia y transmisibilidad a múltiples rangos de hospedadores (Figura 8).
Tres son los factores principales que facilitan que los coronavirus tengan esa increíble capacidad de salto inter-especie:
- La tasa de mutación relativamente alta en la replicación del ARN. En comparación con otros virus de ARNss (monocatenarios), las tasas de mutación estimadas en los coronavirus podrían considerarse de «moderadas» a «altas». Además, la tasa de mutación es a menudo y todavía más alta cuando los coronavirus no están bien adaptados al hospedador. En comparación con el SARS-CoV, con una alta tasa de mutación, la del SARS-CoV-2 es aparentemente menor, lo que sugiere un mayor nivel de adaptación a los seres humanos. Además del SARS-CoV-2, esto también se aplica al MERS-CoV, que se adapta bien a los dromedarios. Los coronavirus se caracterizan por tener una enzima exoribonucleasa que es capaz de corregir errores lo que se traduce en que su tasa de mutación es menor que otros virus ARN, que no lo hacen. Se ha observado que la delección de esta enzima da como resultado una capacidad de mutación y atenuación extremadamente altas o incluso hace al virus inviable.
- El tamaño de su ARN viral. El tamaño grande del ARN en los coronavirus añade una mayor plasticidad en la modificación del genoma para que se den mutaciones y recombinaciones, lo cual resulta en una ventaja para la aparición de nuevos coronavirus cuando las condiciones se vuelven apropiadas.
- Cambios aleatorios. Los coronavirus cambian de forma aleatoria y frecuente durante la replicación de su ARN a través de un mecanismo único llamado «opción de copia», en un animal hospedador que puede servir como «vaso de mezclas». Los ARN de distintos coronavirus podrían recombinarse en una misma célula infectada, generando así nuevos coronavirus. Se han encontrado pruebas filogenéticas de recombinación natural en coronavirus humanos como el HCoV-HKU1 o el HCoV-OC43, así como en coronavirus animales como bat SL-CoV y batCoV-HKU9.
Interacción virus-hospedador en relación con la transmisión.
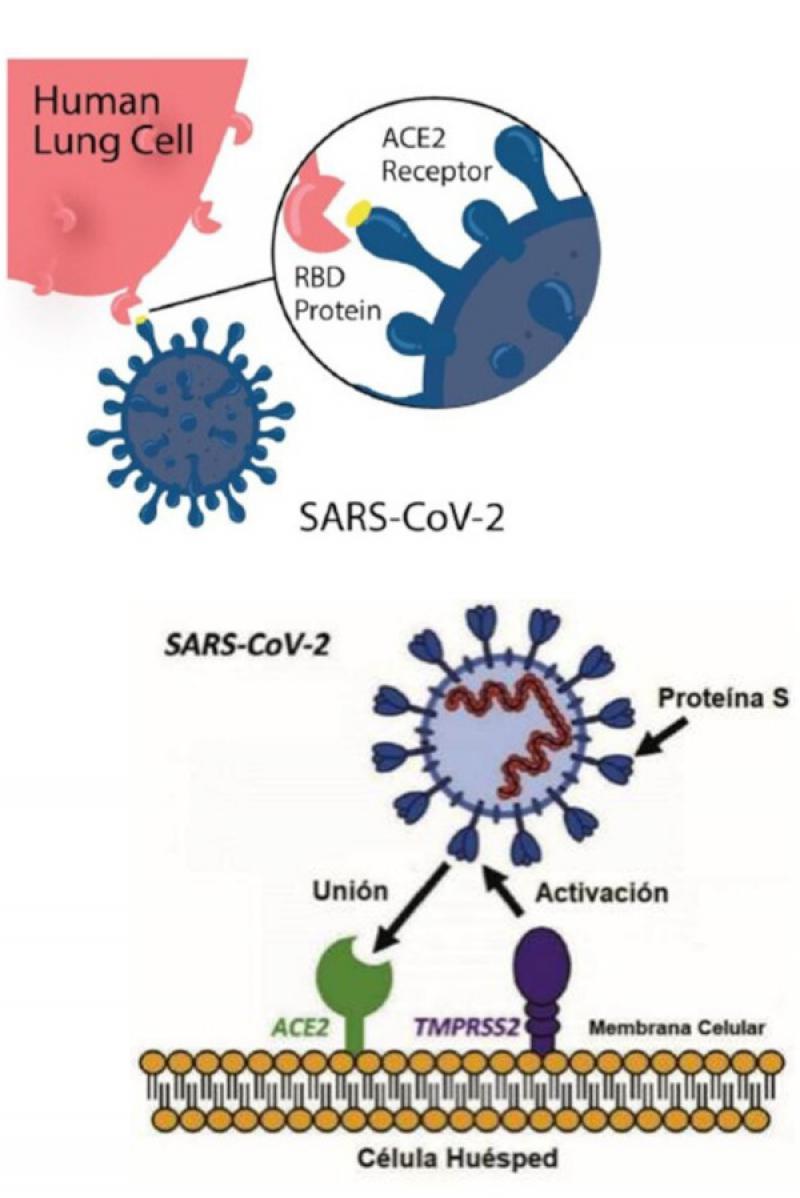
Además de los tres factores virales mencionados anteriormente, la interacción viral con el receptor de las células a las que infecta en el hospedador es otro factor clave que influye en la interacción virus-hospedador. Basado en el análisis comparativo entre aislamientos de diversos coronavirus como el SARS-CoV, se observa como este experimenta una rápida adaptación en diferentes hospedadores, particularmente a través de mutaciones en la región RBD (Receptor Binding Domain) de la proteína S.
El RBD de SARS-CoV y SARS-Cov-2 es capaz de reconocer los receptores ACE2 (enzima convertidora de angiotensina 2) de varios animales donde se incluyen murciélagos, civetas, ratones, perros, gatos, visones y hurones, lo que permite la transmisión del virus entre especies. De hecho, en el SARS-CoV se observó que solo 6 residuos de aminoácidos eran diferentes para el RBD entre los aislados virales de civeta y humanos, 4 de ellos ubicados en el motivo de unión al receptor para la interacción con el receptor ACE2.
También se ha comprobado que existe una diferencia del 30% entre el SARS-CoV y el SARS-CoV-2 en la unidad S1 de la proteína S, lo que implica que la afinidad de unión de la proteína S con la ACE2 humana podría haberse alterado en este último. De hecho, un estudio de criomicroscopía electrónica indicó una afinidad de 10 a 20 veces mayor para el receptor ACE2 en el SARS-CoV-2 versus SARS-CoV. Curiosamente, el coronavirus humano HCoV-NL63 también se une a ACE2, pero por una región diferente de la proteína S. Existen otros receptores para los HCoV, como la aminopeptidasa N para HCoV-229E y el ácido siálico 9-O-acetilado para HCoV-OC43 (Figura 9 y 10).
Patogenia
En condiciones de laboratorio, el periodo de incubación en animales parece ser similar al que se observa en humanos, es decir, entre 2 y 14 días, con una duración media de 5 días. Sin embargo, se requieren estudios adicionales para estimar con fiabilidad la duración media de incubación, el curso clínico y periodo de infecciosidad del virus.
Bajo condiciones experimentales, los animales infectados han mostrado la presencia del virus en el tracto respiratorio y, en algunos casos, lesiones en la tráquea y los pulmones, asociadas con disnea y tos.
Diagnóstico
Diagnóstico clínico
Hasta la fecha, los conocimientos que se tienen acerca de la sensibilidad de diferentes especies animales a la infección por SARS-CoV-2 y los signos clínicos son limitados (Tabla 1). La evidencia actual sugiere que los síntomas pueden incluir tos, estornudos, dificultad respiratoria, descarga nasal, descarga ocular, vómito o diarrea, fiebre y letargia. Y al igual que en los humanos, ocurren infecciones asintomáticas.
Diagnóstico patológico
Se necesitan más estudios para categorizar sistemáticamente los hallazgos post-morten de la infección por SARS- CoV-2 en animales.
En ratones transgénicos que expresan la versión humana del principal receptor del virus, la ACE2, la histopatología revela neumonía intersticial con una infiltración celular inflamatoria significativa alrededor de los bronquiolos y vasos sanguíneos, así como detección de antígenos virales en las células epiteliales bronquiales y alveolares. En hámsteres dorados, se han constatado cambios histopatológicos en el tracto respiratorio y en el bazo. Los macacos Rhesus infectados por SARS-CoV-2 han desarrollado lesiones similares a las observadas en humanos. Por su parte, los gatos jóvenes infectados por SARS-CoV-2 muestran lesiones masivas en el epitelio de la mucosa traqueal y en los pulmones.
Finalmente, en los hurones, el SARS-CoV-2 se puede replicar en el tracto respiratorio superior sin causar enfermedad severa, pero sí desarrollando un cuadro patológico consistente en perivasculitis y vasculitis linfoplasmocíticas graves, incremento de los neumocitos tipo II, macrófagos y neutrófilos en el lumen y en los septos alveolares, y peribronquitis en los pulmones.
Diagnóstico diferencial
Se deberán excluir otras causas de enfermedad respiratoria o digestiva antes de efectuar un diagnóstico asertivo por SARS-CoV-2. Es muy importante comprobar la existencia de una relación epidemiológica con una infección confirmada en humanos o en otros animales y así reducir la lista de patógenos diferenciales.
Diagnóstico laboratorial
Dependiendo del tipo de prueba, se pueden incluir muestras simples o combinaciones de hisopados bucofaríngeos, nasales, rectales y de sangre. Las muestras fecales pueden ser consideradas en situaciones en las que el muestreo directo no es posible debido a los riesgos que representa para el animal o el personal a cargo de las pruebas (p. ej. muestreo en fauna silvestre o animales de zoológico).
Técnicas de diagnóstico
Detección directa del agente:
- Reacción en cadena de polimerasa con transcripción inversa (RT-PCR).
- Amplificación isotérmica mediada por bucle mediante transcriptasa inversa (RT-LAMP).
- Aislamiento del virus.
- Secuenciación del genoma viral.
Técnicas serológicas:
- Prueba ELISA para la detección de anticuerpos.
- Prueba de neutralización viral (VN).
- Otras pruebas para la detección de anticuerpos IgM o IgG.
Prevención y control
- Las medidas de bioseguridad e higiene son esenciales para prevenir la transmisión del SARS-CoV-2 (Figura 11-12).
- Las personas con sospecha o confirmación de infección por SARS-CoV-2 deberán restringir el contacto con animales mamíferos, incluyendo las mascotas, como lo harían con otras personas durante el curso de su enfermedad.
- Los animales con sospecha o confirmación de infección por SARS-CoV-2 deberán permanecer separados de otros animales y humanos.
- Se están utilizando algunas especies animales (p. ej. hurones, gatos, etc.) como modelos experimentales para probar candidatos vacunales para su uso en humanos.
- Algunas vacunas contra el SARS-CoV-2 para uso en visones y otras especies animales susceptibles a la infección por SARS-CoV-2 se encuentran en diferentes etapas de desarrollo y aprobación, no existe ningún tratamiento específico para la COVID-19.
Coronavirus animales y humanos zoonósicos
La pérdida de biodiversidad, la rápida y masiva urbanización, los sistemas agrícolas o el tráfico de animales, entre otros, posibilitan la destrucción de los hábitats naturales de los animales que sirven como reservorios de ciertos virus, maximizando la estrecha asociación de dichos reservorios con los seres humanos. No cabe la más mínima duda de que esto ha conducido a la aparición de nuevas enfermedades zoonósicas producidas por virus como el Ébola, Hendra, Nipah, Hanta y los distintos coronavirus zoonósicos.
La facilitación del contacto del reservorio animal con otras especies animales (hospedadores intermediarios) y con el ser humano, va a facilitar la recombinación genética de distintos coronavirus con la consiguiente aparición de nuevos virus que pueden llegar a afectar de forma importante a las distintas especies animales en general y a la especie humana en particular. Los siguientes son algunos ejemplos de estos coronavirus animales y humanos zoonósicos (Figura 13).
A continuación, vamos a describir algunos de estos coronavirus que afectan a nuestros animales, algunos estaban anteriormente circunscritos a los animales de producción, pero en la medida en que estos animales de producción han pasado a ser uno más de nuestros animales de compañía los hemos incluido, aunque sea brevemente, para que sean conocidos. Por eso que no sorprenda al lector verlos en este artículo sobre «INFECCIÓN POR SARS-CoV-2 EN ANIMALES DE COMPAÑIA»
Coronavirus en perros y gatos
Entre las especies animales que han demostrado mayor sensibilidad al SARS-CoV-2 se encuentran los animales de compañía (perros y gatos). Diversos estudios científicos así lo demuestran.En un artículo de Shen et al. (2020), se valoró la capacidad de la ACE2 de diferentes especies animales (gatos, perros, chimpancés y bovinos). La ACE2 es el receptor celular específico para las espículas proteicas del COVID-19 como hemos visto anteriormente, unión trascendental para que el virus ingrese en la célula hospedadora e inicie la replicación vírica e infección consecuente.
Pues bien, los resultados de este trabajo concluyen en una mayor sensibilidad de perros y gatos a esta unión específica virus-célula; además, se comprueba que estas especies animales pueden transmitir eficazmente el virus a animales previamente no infectados que cohabiten con ellos. El trabajo concluye proponiendo a los gatos como modelo animal para valorar drogas antivirales o posibles candidatos vacunales frente al SARS-CoV-2.
En un segundo trabajo, realizado por investigadores del Instituto Pasteur de París, se testaron 21 mascotas (12 perros y 9 gatos) que vivían en estrecho contacto con sus propietarios (una comunidad de 20 estudiantes de veterinaria), y entre los que 2 de ellos habían resultado positivos para COVID-19 y otros 11 mostraban signos clínicos compatibles con la infección (fiebre, tos, anosmia, etc.). Aunque algunas mascotas presentaron signos clínicos leves indicativos de infección por coronavirus, ninguna de ellas fue positiva a las pruebas de PCR, ni se detectaron anticuerpos mediante ensayos serológicos. Estos hallazgos pueden servir para una mejor comprensión y evaluación del rango de hospedador del SARS-CoV-2 en condiciones de exposición natural.
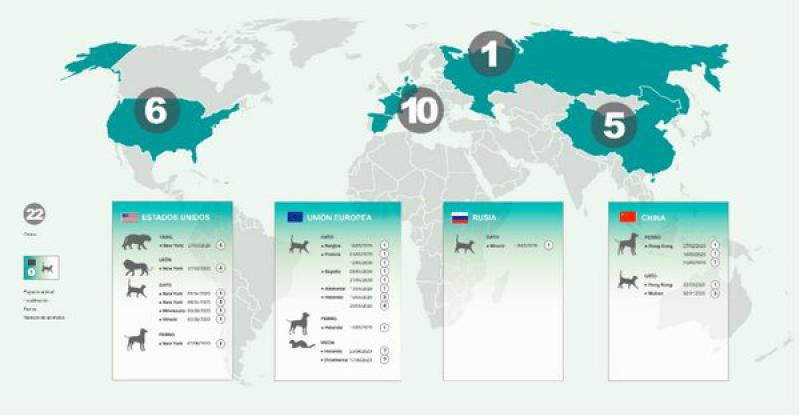
A continuación, vamos a comentar algunos de los casos confirmados de infección por COVID-19 en animales de compañía, y en los que se ha realizado una notificación oficial a la OIE (Figura 14).
El primer trabajo nos confirma un caso de infección por SARS-CoV-2 en perro de raza pastor alemán en Hong-Kong; este ejemplar convivía con una persona COVID (+) que requirió hospitalización. Las muestras recopiladas en el animal a partir de hisopo nasal, faríngeo y rectal, así como las muestras fecales fueron positivas a la PCR.
En el siguiente ejemplo la especie animal afectada es el gato y el país Bélgica. El propietario del animal había sido previamente positivo al virus. El gato desarrolló dificultad respiratoria y diarrea una semana más tarde. Los colegas veterinarios de la Universidad de Liega analizaron las muestras animales (vómito y heces) y detectaron el genoma del virus SARS-CoV-2 por PCR.
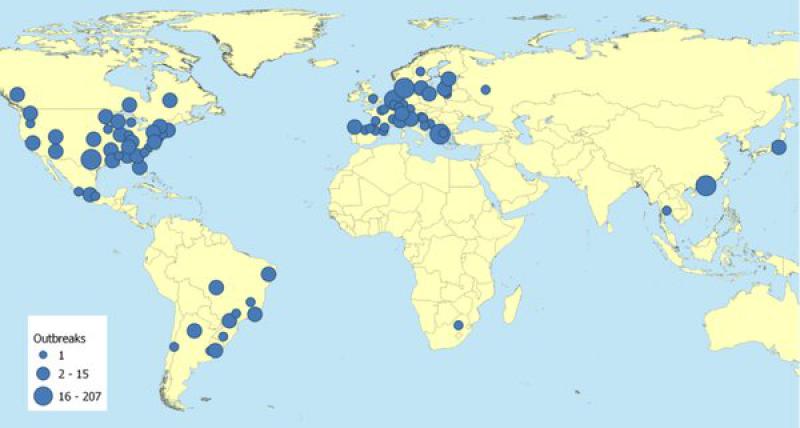
Actualmente (03/06/2021), los estudios científicos han notificado un total de 12 casos de perros infectados por SARS-CoV-2 en todo el mundo. En el caso de los gatos, se han descrito hasta la fecha un total de 18 casos positivos en todo el mundo; al igual que en el caso de los perros, todos ellos en estrecho contacto con personas COVID positivas. La mayoría de estos animales infectados (PCR y/o anticuerpos específicos) si mostraron signos de tipo respiratorio y digestivo. La distribución geográfica y cronológica de estos casos, tanto en perros como en gatos, aparece recogida en la Figura 15.
Coronavirus caninos
Los perros tienen sus propios coronavirus (CCoV), encontrándose dos Alphacoronavirus: los coronavirus entéricos caninos I y II (CCoV-I y CCoV-II), y un Betacoronavirus: el coronavirus respiratorio canino (CRCoV). CCoV-I y CCoV-II, presentan un 96% de identidad de secuencia de nucleótidos en el genoma viral, pero son muy divergentes en el gen de la proteína S. El CCoV-II tiene dos subtipos: el CCoV-IIa clásico y el recombinante CCoV-IIb. Una cepa de CCoV-IIa hipervirulenta y denominada CCoV pantrópica (pCCoV), se aisló en Italia en 2005. Esta variante se asocia con una enfermedad mortal de los perros, caracterizada por leucopenia, gastroenteritis y lesiones graves en los órganos principales.
Actualmente no existen herramientas de diagnóstico capaces de diferenciar las cepas pantrópicas de los CCoVIIa entéricos, ya que están estrictamente relacionados a nivel genético y antigénico y ningún marcador específico de patogenicidad ha sido descubierto hasta ahora. Por lo tanto, la identificación de pCCoV se basa en la detección de CCoV-IIa en tejidos extraintestinales. La infección simultánea de CCoV-I y II puede favorecer la recombinación del genoma viral. El Virus de la Gastroenteritis Transmisible porcino (TGEV) presenta una alta analogía con los CCoV, manteniéndose la hipótesis de que este virus se originó a partir del CCoV-II.
Descubierto en 2003 en el Royal Veterinary College de Londres, el coronavirus respiratorio canino (CRCoV) es un Betacoronavirus que se incluye como agente etiológico dentro del complejo respiratorio canino (tos de las perreras). Por lo general causa signos clínicos leves de tos persistente y secreción nasal. Es un virus altamente infeccioso y es más prevalente en refugios. Desde un punto de vista genético se encuentra estrechamente relacionado con los Betacoronavirus humano HCoV-OC43 y bovino (BCoV).
Coronavirus felinos
Los gatos y otros félidos salvajes, como no podía ser menos, también se ven afectados por ciertos coronavirus (FCoV) incluidos dentro de los Alphacoronavirus, que llegan a ser muy importantes a nivel epidemiológico y clínico. Se han descrito dos tipos diferentes de FCoV, el FCoV-I y FCov-II con una distribución epidemiológica distinta, y características biológicas y moleculares que prueban la diversidad de estos agentes.
El FCoV tipo I se considera el ancestro del linaje felino. Es el más común de los dos tipos y actualmente se encuentra circulando en los reservorios de gatos domésticos a nivel mundial. En contraste, se ha sugerido que el FCoV-II surgió por eventos de recombinación entre coronavirus felinos y caninos. Aunque normalmente se considera una infección entérica, los FCoV pueden tener una distribución más sistémica en su hospedador (incluso en el tracto respiratorio), antes de que las mutaciones críticas en el genoma viral resulten en la adquisición de tropismo por los macrófagos y la progresión a peritonitis infecciosa (PIF).
La PIF puede dar lugar a un cuadro efusivo («húmedo») o no efusivo («seco») a menudo acompañado de lesiones granulomatosas extensas. Esta compleja enfermedad se complica aún más por el fenómeno ADE (Antibody Dependent Enhancement), mediante el cual algunos de los anticuerpos producidos durante la infección pueden funcionar como anticuerpos «Caballo de Troya», facilitando aún más la infección de los macrófagos, conduciendo este a una más rápida y grave progresión de la enfermedad.
Coronavirus roedores y conejos
Se han descrito otros coronavirus que producen infecciones en roedores como el de la sialodacroadenitis de la rata, la hepatitis y la encefalomielitis murina y otros que afectan a conejos produciendo miocarditis y enteritis en esta especie animal. Los coronavirus no conocen límites en cuanto a su capacidad para infectar a numerosas especies animales.
Coronavirus en hurones
En el caso de los hurones, la sensibilidad y transmisibilidad intra-especie parece particularmente relevante y así lo demuestran algunos trabajos científicos. En un manuscrito recientemente publicado en la revista Science autores chinos han investigado la sensibilidad de hurones y animales en estrecho contacto con humanos al SARS-CoV-2, encontrando que este virus se replica escasamente en cerdos, gallinas y patos, a diferencia de hurones y gatos; los gatos fueron especialmente sensibles a la infección vía aerógena. Este estudio revela importantes utilidades en modelos animales para el SARS-CoV-2 así como el manejo de animales para el control de la infección (Jianzhong Shi et al., 2020).
Por su parte, Young-ill Kim et al, en la República de Corea, constatan la infección y rápida transmisión del SAR-CoV-2 en hurones, concluyendo que son especies animales altamente sensibles a la infección que puede ser vehiculada de forma directa e indirecta a través de secreciones nasales, saliva, orina y heces. De nuevo, un magnífico modelo animal para el estudio de ensayos clínicos para nuevos fármacos humanos y candidatos vacunales.
Infección en visiones
En abril de 2020 se describió la primera notificación de SARS-CoV-2 en Países Bajos en visones; posteriormente la infección de detectó en trabajadores de granjas de esta especie animal, confirmándose de esta forma la transmisión humana a visón y viceversa. Desde entonces, infecciones en visones se han detectado en Dinamarca, Italia, España, Suecia y EE. UU. (WHO, 2020).
A finales de abril de 2020, las autoridades de los Países Bajos informaban que 4 granjas de visones habían sufrido brotes por SARS-CoV-2 en varios ejemplares de visones tras mostrar diversos síntomas, fundamentalmente de índole respiratorio, desarrollando una típica neumonía intersticial aguda (Jan Molenaar. et al., 2020). Algunos animales fueron asintomáticos y las tasas de morbilidad y letalidad fueron bajas.
Desde ese momento, el Ministerio de Agricultura, Naturaleza y Calidad de los Alimentos, solicitó una investigación para conocer el origen de las infecciones, que se consideraba de naturaleza humana, ya que varios trabajadores de las granjas habían padecido previamente la enfermedad.
En algunos casos las granjas tenían el mismo propietario, pero en otros casos no se pudo establecer ningún vínculo epidemiológico, por lo que personas que visitaban las diferentes granjas pudieron ser la fuente original, pero también los gatos asilvestrados que cohabitan en ellas o quizás la fauna silvestre. De hecho, en el transcurso de las investigaciones, en las que participó la Universidad de Utretch, se detectaron gatos infectados en las diferentes granjas.
Oreshkova et al. (2020), describieron la enfermedad respiratoria y la mortalidad de visones en dos granjas afectadas. En ambas al menos un trabajador padeció síntomas asociados a SARS-CoV-2 antes de los brotes en los animales. Los estudios genéticos del genoma viral de los visones, demostró la transmisión directa entre visones de una misma granja, pero no la transmisión entre diferentes granjas. Este estudio concluye que el polvo inhalado y cargado de partículas víricas explica la posible exposición de los trabajadores; de hecho, al comparar los virus aislados a partir de diferentes visones y personas infectadas en una misma granja se demostró la similitud genética. Los autores confirmaron la transmisión vía animal-humano en dos de los trabajadores y, por tanto, ha sido la primera descripción de transmisión animal-humano, excepción hecha del primer caso originado en Wuhan (China).
Por otra parte, la clara separación filogenética entre los casos humanos relacionados con granjas y la población general dentro de la misma área geográfica, proporciona otra prueba más de que los animales eran la fuente más probable de la infección. Si bien, aunque los investigadores señalan que encontraron secuencias genéticas en humanos que coinciden con las de visones en varias granjas, apuntan a que no todas estas pueden considerarse transmisiones zoonósicas directas. Así, infecciones humanas posteriores pueden haberse originado a partir de infecciones zoonósicas adicionales o de transmisión doméstica persona a persona (Reusken et al., 2020).
Finalmente, Oude Munnink et al. (2020), realizan una profunda investigación en Países Bajos basada en la técnica de secuenciación genómica (WSG) aplicada específicamente en 16 granjas de visones afectados por SARS-CoV-2, así como a humanos que cohabitaban o trabajaban en ellas. Los resultados de este reciente trabajo concluyen que el SARS-CoV-2 fue introducido desde los humanos diseminándose posteriormente entre los visones. Inmediatamente se implantaron medidas de bioseguridad, vigilancia y sacrificio sanitario de las granjas infectadas (stamping out); aunque se demostró la transmisión a otras granjas sin haberse conocido el modo de transmisión.
El 68% de las personas que cohabitaban en dichas granjas, trabajadores o personas que habían tenido contacto con los animales afectados mostraron evidencias de infección frente al SARS-CoV-2. Los resultados de esta técnica diagnóstica, de altísimo poder de discriminación, han demostrado la infección en personas a partir de cepas de origen animal, proporcionando evidencia científica de la transmisión animal-humano de SARS-CoV-2 en las granjas de visones.

Por otra parte, el 10 de noviembre de 2020, saltó la noticia de que casi 400 casos de COVID-19 en humanos podrían estar vinculados a visones enfermos en granjas peleteras de Dinamarca (ECDC, 2020). La mayoría de los casos humanos y animales notificados se han ubicado en la región geográfica de North Jutland. Para prevenir el denominado fenómeno conocido como «derrame inverso» que describe la transmisión de un virus desde humanos a animales, las autoridades danesas decretaron el sacrificio de 17 millones de visones de las casi 1 200 granjas peleteras del país como medida cautelar (Figura 16).
La decisión se tomó a raíz de los hallazgos que sugerían que la cepa del virus que circulaba entre visones y humanos podría haber mutado lo suficiente para afectar a la eficacia de las futuras vacunas, de ahí la necesidad de tomar medidas inmediatas. Dinamarca informó de que había descubierto visones enfermos en sus granjas peleteras en junio, pero en aquel momento parecía que los trabajadores humanos eran los que transmitían el virus a los visones.
Esto nos sugiere que la transmisión de visones a humanos y viceversa es más común de lo que se pensaba. Los visones podrían ser más susceptibles al SARS-CoV-2 que otros animales debido a una combinación de factores genéticos y ambientales. Los visones de granjas no presentan mucha diversidad genética, lo que puede favorecer la transmisión y la susceptibilidad a las enfermedades infecciosas. Asimismo, los visones de las granjas suelen vivir en densidades relativamente altas, lo que favorece la propagación del virus. El virus transmitido a los visones por humanos se ha adaptado a infectar a estos animales habiendo experimentado cuatro mutaciones.
A este virus se le conoce como variante 5, pues se han detectado otras cuatro versiones del coronavirus que también circulaban entre visones. Esta nueva variante 5 ha vuelto a saltar a humanos, probablemente trabajadores de las granjas. Las mutaciones de esta variante 5 afectan sobre todo a la proteína S, que es esencial para que el SARS-CoV-2 pueda infectar, siendo también la diana de muchas de las vacunas que están en desarrollo. Aunque la posibilidad de que estos cambios puedan disminuir la efectividad de las vacunas es remota, un estudio realizado por el Instituto Serológico de Dinamarca ha concluido que los anticuerpos neutralizantes procedente de humanos que han pasado la infección no neutralizan tanto a esta nueva variante 5 como lo hace frente a «cepas convencionales».
Por el momento las observaciones iniciales de la OMS indican que el cuadro clínico inicial, la gravedad y la transmisión entre las personas infectadas son similares a los de otros virus SARS-CoV-2 circulantes. Esta variante presenta una combinación de mutaciones o cambios que no se han observado previamente. Las conclusiones preliminares de la OMS indican que esta variante concreta asociada a los visones y detectada tanto en estos animales como en humanos presenta una sensibilidad moderadamente disminuida a anticuerpos neutralizantes. Sin embargo, se requieren más estudios científicos y de laboratorio para verificar las conclusiones preliminares.
Como corolario, en España, un brote de coronavirus obligó a acabar con los casi 100 000 de animales de una granja de La Puebla de Valverde, en Teruel, por las mismas razones esgrimidas anteriormente.
Coronavirus infección en animales de zoológico
En el contexto de animales de zoológico, un día de abril de 2019 nos despertamos con la noticia que el SARS-CoV-2 había contagiado a una tigresa malaya de 4 años llamada Nadia en Nueva York (NY). Se tomaron muestras de cavidad nasal y faringe; para esta prueba lógicamente el animal fue sedado; los resultados fueron positivos para COVID-19. Pero Nadia no fue el único caso en el recinto. Los veterinarios habían observado que otros grandes felinos también tosían; de hecho, la infección afectó a varios ejemplares de tigres de Amur y leones africanos; se les hizo un análisis de heces, por cierto, prueba que evita tener que sedar a los animales y también fueron positivos. Según fuentes del zoo, los tigres y leones se han recuperado, tosen menos y han vuelto a comer con normalidad.
Pues bien, los estudios han concluido que los felinos fueron contagiados por un trabajador del zoo de Nueva York. El individuo no sabía que portaba el virus. Era asintomático en el momento de la transmisión efectiva y parece que no ha desarrollado la enfermedad clínica a posteriori. Las instalaciones del zoo han permanecido cerradas desde el mes de marzo cuando comenzó la pandemia, y lógicamente el zoo de NY ha puesto en marcha un protocolo de seguridad para sus trabajadores para evitar futuros contagios.
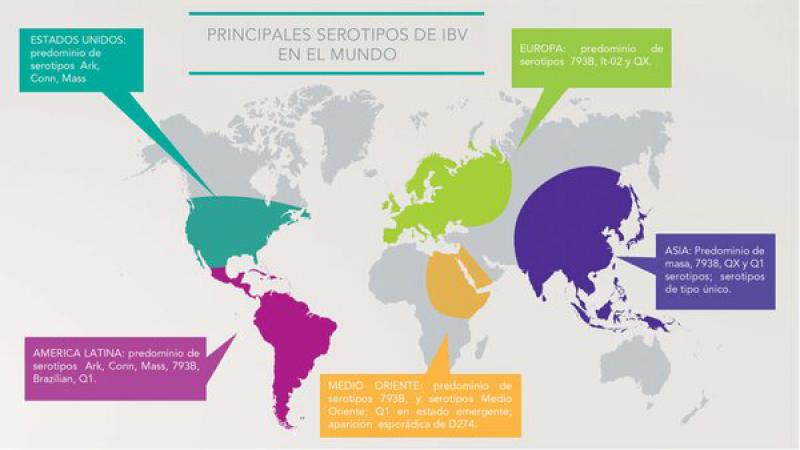
Coronavirus aviares: virus de la bronquitis infecciosa aviar (IBV)
Lo que las gallinas nos enseñan sobre el coronavirus y su control
Descrito en 1931, el coronavirus que afecta a las aves de corral y que causa enfermedades respiratorias en los pollos, Bronquitis Infecciosa Aviar (IBV), pertenece al grupo de los Gammacoronavirus aviares. Los virus aviares del grupo de los Gammacoronavirus no infectan ni causan enfermedades en los humanos. El virus COVID-19 está en el grupo de los virus Betacoronavirus junto con el SARS-CoV y el MERS-CoV (Figura 17).
El IBV es el coronavirus más común y extendido en todas las granjas aviares del mundo, produciendo cuadros de bronquitis infecciosa. Este virus es responsable de una morbilidad y mortalidad significativas en aves de corral, incluidos faisanes, pavos y gallinas de Guinea de todas las edades. Son especialmente las aves más jóvenes las más susceptibles, lo que causa pérdidas económicas masivas.
En las aves el virus puede penetrar a través de diferentes rutas, como la vía feco-oral o la aérea. El IBV generalmente se replica en las células epiteliales ciliadas del tracto respiratorio, causando cuadros respiratorios graves con tos, secreción nasal, estertores, jadeo e inflamación de los senos nasales. Después de infectar a las células del tracto respiratorio, se genera una viremia secundaria (presencia del virus en sangre) llegando a otros órganos donde puede producir lesiones. Así, el IBV es capaz de producir trastornos del tracto digestivo, reproductivo y urogenital, lo que explica la nefritis y el síndrome de caída de la puesta descrito en esta infección.
El IBV se agrupa en seis genotipos genéticamente divergentes (GI, GII, GIII, GIV, GV, GVI) con 32 linajes. Solo el genotipo I agrupa veintisiete linajes. Cada uno de los seis genotipos restantes contiene un solo linaje. Algunos de estos linajes se distribuyen en varios continentes, países o regiones, mientras que otros son autóctonos de determinadas zonas geográficas. Cada linaje comprende diferentes virus, cada uno denominado cepa y con una designación específica (por ejemplo, Italy 02, QX, Arkansas…).
El IBV está en continua evolución. La secuenciación de genes y el análisis filogenético de cepas de virus sugieren la aparición de nuevas cepas a través de mecanismos de mutación y recombinación genética a partir de una cepa ancestral (Massachusetts IBV). Es más que probable que dichos cambios genéticos se hayan dado por la presión inmunológica ejercida a través de la infección natural o por el uso de vacunas vivas atenuadas, además de por infecciones mixtas frecuentes.
La proteína S1 es el principal componente antigénico del virus, responsable de inducir anticuerpos neutralizantes e inmunidad protectora. Los epítopos de diferentes cepas pueden variar su composición de aminoácidos, lo que puede modificar la forma tridimensional de la proteína y, por lo tanto, la inmunidad inducida por una cepa puede no proporcionar suficiente protección cruzada frente a otras.
La inmunidad local de la mucosa respiratoria juega un papel primordial en la protección de las aves frente al IBV. La respuesta inmunitaria humoral de mucosas, a través de la formación de anticuerpos locales (principalmente de clase IgA), neutralizan el virus e inhiben su unión a las células epiteliales. Las glándulas de Harder y lagrimales producen IgA local, mientras que la respuesta de inmunidad celular de tipo Th1, en la que intervienen linfocitos CD8 + citotóxicos y CD4 + activados, juegan un papel determinante en la eliminación de la infección del tracto respiratorio y en control de su diseminación al resto de órganos diana. En cuanto al papel protector de la inmunidad humoral a base de anticuerpos circulantes, es un asunto que sigue en controversia.
Varios estudios han demostrado que los títulos de anticuerpos circulantes no se correlacionan con la protección del tracto respiratorio (no son correlato de protección). A pesar de estos estudios, otros han demostrado que los altos niveles de anticuerpos circulantes en las gallinas ponedoras son extremadamente importantes para proteger contra la infección del tracto reproductivo, la disminución de la producción de huevos y el deterioro de la calidad de la cáscara del huevo (Figura 18).
Los anticuerpos circulantes (principalmente IgG) inducidos por las vacunas limitan la propagación del virus desde el tracto respiratorio a otros órganos susceptibles, incluidos los riñones y el tracto reproductivo. Las vacunas vivas atenuadas administradas por aspersión estimulan la inmunidad humoral y celular local.
Ninguna vacuna proporciona una protección completa contra las diferentes variantes. Aunque la protección cruzada puede ser de bajo nivel, se ha demostrado que ciertas vacunas pueden proporcionar una mejor protección parcial que otras contra un virus de campo en particular. En condiciones de campo, a menudo se usa una combinación de dos o más tipos de vacunas contra el IBV para maximizar la protección cruzada. Esta protección cruzada parcial del virus vacunal heterólogo puede deberse a la unión de anticuerpos neutralizantes a epítopos conservados en la proteína S1 (Figura 19).
Otros coronavirus aviares del género Deltacoronavirus incluyen el Bulbul-CoV- HKU11, Wigeon-Cov-HKU20, White eye-CoV-HKU16, Common Moorhen-CoV-HKU21, Munia- CoV-HKU13 y Night heron-CoV-HKU19, que pueden infectar gravemente a las aves. Aunque ha habido diferentes intentos para el desarrollo de una vacuna eficaz contra los distintos coronavirus aviares, las mutaciones rápidas y las variaciones antigénicas son un obstáculo para desencadenar con éxito la respuesta inmunitaria local o de mucosas y poder prevenir la enfermedad.
Coronavirus porcinos
Los cerdos se ven afectados por coronavirus de los géneros Alpha-, Beta- y Deltacoronavirus. Actualmente son seis los coronavirus que circulan en la población porcina. La infección por Alphacoronavirus en cerdos incluye cuatro especies virales: el coronavirus Respiratorio Porcino (PRCoV-1984), el virus de la Gastroenteritis Transmisible (TGEV-1946), el virus de la Diarrea Epidémica Porcina (PEDV-1977) y el coronavirus del Síndrome de Diarrea Aguda Porcina (SADS-CoV). La infección por Deltacoronavirus en cerdos incluye el Deltacoronavirus porcino (PDCoV-2009). El Betacoronavirus responsable de la enfermedad porcina es el virus de la Encefalomielitis Hemaglutinante Porcina (PHEV-1962). Los PRCoV causan infecciones respiratorias, mientras el TGEV, PEDV, SADS-CoV y PDCoV causan gastroenteritis aguda y PHEV induce trastornos neurológicos y digestivos graves.
El TGEV es el coronavirus porcino más antiguo conocido. Descrito en 1946, alcanzó su mayor prevalencia entre las décadas de 1970 a 1990, tanto en Europa como en EE. UU., siendo más del 95 % de las granjas europeas seropositivas en los años 80. Es un virus altamente infeccioso causante de diarrea, deshidratación y ocasionalmente vómitos como hecho diferencial, provocando una elevada mortalidad en cerdos jóvenes. El virus GET está completamente secuenciado y se conoce un solo serotipo. Presenta un alto nivel de homología con coronavirus bovinos y humanos, así como con el PRCoV.
De hecho, el PRCoV se trata de una variante atenuada de TGEV, originada por la detección de la región 5' en el gen que codifica para la proteína S del virus, siendo un ejemplo de la evolución de coronavirus con propiedades de virulencia y tropismo alterados. Desde el punto de vista epidemiológico, se describen cuadros epidémicos y endémicos. La vía de contagio es la oro-fecal, aunque las cerdas reproductoras pueden transmitir el virus por la leche. El virus puede persistir en los cerdos tras la desaparición de los signos clínicos y puede ser excretado vía heces durante 10 semanas, por lo tanto, muchos animales quedan como portadores asintomáticos durante este tiempo, produciendo nuevas infecciones en animales susceptibles.
El PRCoV tiene una incidencia y prevalencia muy baja a nivel mundial, con un impacto económico leve sobre la producción porcina. Como ya se ha comentado, este virus es una variante de la familia del virus de la GET que infecta el tracto respiratorio, y que no se elimina vía heces. El virus infecta animales de todas las edades, bien por contacto directo o mediante transmisión aérea, siendo más prevalente en áreas de alta densidad porcina. La presentación de los signos clínicos es mayormente subclínica, por lo que podemos encontrar muchos animales seropositivos sin ninguna clínica en la mayoría de los países del mundo de forma endémica. Tras la infección, el tiempo de excreción nasal del virus es de unos 7-15 días. Su prevalencia es estacional, aumentando en los meses fríos y reduciéndose en los cálidos. Actualmente, la seroprevalencia en España es muy baja.
Perteneciente al género Betacoronavirus, el PHEV es un virus que produce trastornos a nivel del tracto digestivo y nervioso en cerdos de todas las edades. Fue aislado por primera vez en 1962 e identificado como un virus neurotrópico que mostró presencia subclínica en todo el mundo. Está ampliamente difundido en la población de cerdos de EE. UU. y Europa, pero la enfermedad clínica es infrecuente. Esto es debido a que la mayoría de las cerdas han sido infectadas, son inmunes y pasan los anticuerpos a sus lechones a través del calostro, lo que los protege durante el periodo en que son vulnerables. Las cepas varían en virulencia, dando lugar a dos síndromes diferentes: la enfermedad del vómito más desmedro y la encefalomielitis.
El PEDV es un virus entérico importante de los cerdos que se introdujo en la población porcina a principios de la década de 1970. Afecta principalmente a los cerdos en crecimiento y engorde y se cree que es consecuencia de la propagación de los murciélagos y el consiguiente incremento del contacto entre ambas especies animales. Se conoce un solo serotipo. La virulencia de los virus aislados en diferentes países durante los cuadros clínicos fue similar, con una mínima dispersión genética. El virus se diseminó de forma epidémica por toda Europa en la década de los 70, originando graves cuadros diarreicos en lechones lactantes y diarreas recurrentes en lechones destetados y cerdos de cebo. La morbilidad en cualquier fase de producción puede llegar al 100 %, siendo más variable en las cerdas reproductoras.
La mortalidad en lechones lactantes puede llegar al 100 %, siendo muy «normales» tasas de entre 30% y 50%. Nuevos brotes epidémicos, tanto en Europa como en Asia, tuvieron lugar en los años 80, 90, 2000 y hasta el 2009 (Italia, Corea, Tailandia), con cuadros intermitentes en gran parte de los países productores de porcino a nivel mundial.
El SADS-CoV, es un Alphacoronavirus porcino causante de enteritis en lechones y que tiene más del 86 % de homología de secuencia con los Alphacoronavirus de murciélago HKU2-CoV, lo que indica que SADS-CoV y HKU2-CoV descendieron de un ancestro común. Se acaba de publicar recientemente un trabajo que demuestra que el SADS-CoV puede infectar células humanas in vitro.
En 2012, se detectó en cerdos de Hong Kong un nuevo coronavirus, el PDCoV. Está estrechamente relacionado con el Deltacoronavirus UAE-HKU30 de la codorniz. Se propuso que el virus se originó a partir de la recombinación entre coronavirus de gorriones CoV HKU15 y Bulbul-CoV-HKU11 con cambio de hospedador entre coronavirus aviares y mamíferos. Afortunadamente solo causa una infección leve en los cerdos.
En infecciones experimentales, también se ha podido comprobar que los cerdos pueden infectarse con el MERS-CoV y el SARS-CoV, pero no con el SARS-CoV-2, aunque su importancia a nivel epidemiológico es insignificante, no habiéndose descrito hasta ahora infecciones en humanos procedentes de coronavirus porcinos.
Coronavirus de rumiantes
En cuanto a los rumiantes, los coronavirus bovinos (BCoV) son conocidos de antiguo. Perteneciente al género Betacoronavirus, tienen la capacidad de causar graves trastornos clínicos, que van desde enfermedades entéricas, como enteritis en terneros recién nacidos y enteritis hemorrágica en bovinos adultos, a una forma respiratoria dentro del síndrome plurietiológico conocido como fiebre del transporte en bovinos, y que afecta a todos los grupos de edad.
Se cree que los BCoV evolucionaron a partir de coronavirus de roedores. Se han descrito varios coronavirus de tipo bovino en otros animales domésticos como ovejas, cabras, búfalos de agua, llamas, alpacas y algunos rumiantes salvajes como venado sambar, venado de cola blanca, venado sika, venado de agua, caribú y alce. También se encuentran tipos similares de virus en varias especies de antílopes, bisontes, jirafas y dromedarios (Figura 20).
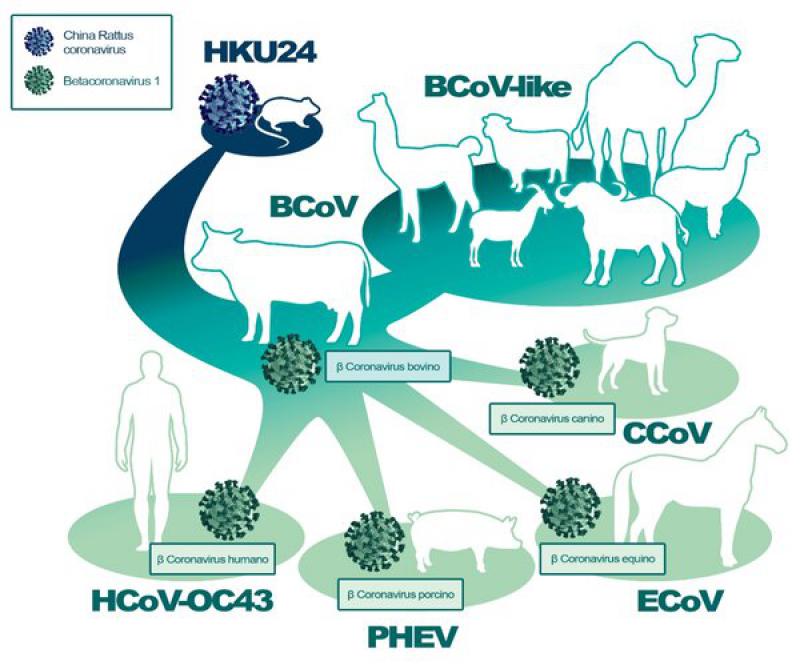
Coronavirus equinos
El coronavirus equino (ECoV), descrito por primera vez en 1999 en muestras fecales de caballos en Carolina del Norte (EE. UU.), es el único coronavirus descrito en esta especie animal, estando presente especialmente en potros de menos de 2 semanas de edad. Se cree que el ECoV es descendiente de los BCoV y son responsables de causar enteritis autolimitante en la población equina. Aunque los équidos no se infectan naturalmente por MERS-CoV, un estudio llevado a cabo en Egipto mostró que en 3 de 42 burros se detectó ARN de MERS-CoV en muestras respiratorias de estos animales.
La amenaza de emergencia de nuevos coronavirus humanos (HCoV)
Para finalizar este repaso a la infección por SARS-CoV-2 y su relación con los animales de compañía, nos quedaría hacer una breve especulación sobre la posibilidad de aparición de nuevos HCoV. En este sentido, la amplia diversidad de coronavirus en murciélagos ofrece enormes oportunidades para la emergencia de nuevos HCoV. En este sentido, estos coronavirus quirópteros sirven como acervo genético de los HCoV, constituyéndose en una importante «reserva viral» para la aparición de nuevos coronavirus que sean capaces de infectar a humanos.
Tanto sus tasas de mutación como su alta capacidad de recombinación genética impulsan la evolución de estos virus y su posible salto entre especies. Tanto la adquisición como la pérdida de genes de codificación proteica tienen el potencial de modificar drásticamente los fenotipos virales. Entre las proteínas accesorias del SARS-CoV, se ha pensado que ORF8 es importante en la adaptación a los seres humanos, ya que se aislaron virus de murciélago relacionados con el SARS-CoV, que codificaban proteínas ORF8 divergentes sin capacidad de adaptación a infectar células humanas.
En este mismo virus de murciélago relacionado con el SARS-CoV se encontraron detecciones de 29 nucleótidos características del SARS-CoV de cepas aisladas al comienzo de la epidemia humana de 2002-2003. Esta detección divide a ORF8 en ORF8a y ORF8b y se cree que es una mutación adaptativa que promueve el cambio de hospedadores. Asimismo, se ha demostrado que la epidemia MERS-CoV surge a partir de eventos de recombinación entre diferentes linajes, lo que ocurrió en dromedarios en Arabia Saudita. También la propia presión del sistema inmunitario del hospedador puede hacer que se den cambios en los genomas virales. Un ejemplo de este efecto de presión inmunológica es la pérdida de un ORF4 en la cepa prototipo de HCoV-229E. Por último, pero no menos importante, es que la evolución de los nuevos HCoV también está impulsada por la presión de selección en sus reservorios. Parece que los murciélagos están bien adaptados a los coronavirus anatómica y fisiológicamente.
Así, el bajo grado de respuesta proinflamatoria que experimentan los murciélagos frente a los coronavirus reduce eficazmente la patología desencadenada por estos, haciendo que estos animales no muestren apenas sintomatología asociada a la infección y por supuesto mortalidad asociada a la misma. Es por ello por lo que los CoV no son patógenos o causan síntomas leves en sus reservorios, ya sean estos murciélagos o camellos. El bajo nivel de respuesta frente a la infección por parte de estos animales hace que el virus se replique de forma importante sin provocar una fuerte respuesta inmunitaria. Esto podría explicar el «gran misterio» del porqué de la existencia de portadores asintomáticos y qué es verdaderamente lo que causa la gravedad de la infección humana.
Como ya sabemos, los cuadros graves producidos por estos virus se deben fundamentalmente a la hiperactivación de la respuesta inmunitaria con el desarrollo de la tan ya conocida «tormenta de citocinas». Tanto es así, que actualmente se establecen parámetros inmunológicos (como por ejemplo la medición de los niveles de interleucina-6, entre otros), que son capaces de establecer un pronóstico de mayor o menor gravedad. Por el contrario, en los portadores asintomáticos esta respuesta inmunitaria se desarrolla bajo un estricto control de la inflamación mientras el virus lleva a cabo su replicación, siendo sobre todo la respuesta de inmunidad innata, con la producción de interferón de tipo I, la que controla la replicación y diseminación viral en primer término, pudiendo llegar a aclarar la infección de forma rápida.
Esta respuesta basada en la producción de interferón es particularmente potente en los murciélagos. Además, la actividad de las células Natural Killer en murciélagos se encuentra suprimida frente a estas infecciones. Esto hace que su capacidad de reservorio principal sea muy elevada. Por otra parte, el alto nivel de especies reactivas de oxígeno (ROS) generado por la alta actividad metabólica de los murciélagos puede suprimir la replicación de virus en general y de los coronavirus en particular, afectando en estos últimos la capacidad de la enzima exoribonucleasa, proporcionando así la presión de selección para la generación de cepas de virus altamente patógenas cuando se introducen en un nuevo anfitrión. Las cepas de CoV más patógenas también podrían evolucionar por recombinación, lo que llevaría a la adquisición de nuevas proteínas o características proteicas para su adaptación al hospedador. Visto así no es casualidad que hayan surgido tres nuevos HCoV en las últimas dos décadas.
El surgimiento del SARS-CoV-2 sigue el mismo patrón general que siguieron otros coronavirus como el SARS-CoV y el MERS-CoV. Aunque quedan muchas dudas acerca de su origen, reservorio animal, hospedador intermediario y otras más, de lo que estamos seguros es de que no existe la más mínima evidencia de que el SARS-CoV-2 haya sido una «creación humana», liberado a la población ya sea de forma accidental o deliberada en un acto de bioterrorismo internacional.
Todo lo anterior, creo que también ha puesto en valor de manera fehaciente la dilatada experiencia de los veterinarios en el estudio, diagnóstico y prevención de los distintos coronavirus animales, con los que venimos enfrentándonos desde 1931 en que se describió el primero, la Bronquitis Infecciosa Aviar (IBV).
En definitiva, los coronavirus y su relación con los animales y los humanos no dejan de sorprendernos…Y LO QUE NOS QUEDA.
Información adicional
El Centro para el Control de Enfermedades (CDC) ubicado en Atlanta USA, tiene a nuestra disposición un enlace en el que se informa de forma actualizada de esta pandemia con relación a los animales. De esta forma y a través de su enlace se puede acceder a informaciones diversas. Por ejemplo: ¿Puedo contraer el COVID-19 a partir de mi mascota? ¿pueden los animales portar el virus en su piel o pelo? ¿qué animales pueden tener el virus? ¿pueden los animales silvestres transmitir el virus a las personas o mascotas?
Asimismo, la Organización Mundial para la Sanidad Animal (OIE), tiene una página específica sobre COVID-19 y animales, con preguntas y respuestas y a la que podéis acceder mediante el enlace mostrado en la Figura 22.
Finalmente añadimos una infografía recientemente publicada por el Consejo General de Colegios Veterinarios de España, y en la que podéis visualizar a modo de resumen lo que sabemos sobre el COVID-19 y los animales de compañía, así como las medidas preventivas que debemos cumplir los veterinarios (Figura 23).


Conclusiones
- En la actualidad no hay evidencia científica que sugiera que los animales domésticos infectados por humanos tengan un papel epidemiológico relevante en la propagación del SARS-CoV-2. Por tanto, no existe justificación alguna para implementar medidas relacionadas con los animales de compañía (perros y gatos) que puedan afectar a su bienestar (Figura 24).
- Los casos en el hombre se deben fundamentalmente a contactos de persona a persona, a excepción de los casos descritos en Holanda y Dinamarca en granjas de visones.
- Se ha constatado la vía de transmisión de personas infectadas a animales susceptibles (perros, gatos, visones, tigres/leones) (antropozoonosis).
Como corolario, queremos enfatizar de nuevo la importancia de este binomio «animal de compañía y salud pública», con especial atención a las zoonosis emergentes como el SARS-CoV-2 con el que tendremos que estar especialmente atentos a nuevas publicaciones y nuevos descubrimientos en el contexto de la sanidad animal.
Bibliografía
- Ahn M, Anderson DE, Zhang Q, Tan CW, Lim BL, Luko K, et al. Dampened NLRP3-mediated inflammation in bats and implications for a special viral reservoir host. Nat Microbiol. 2019; 4: 789-99.
- Binn LN, Lazar EC, Keenan KP, Huxsoll DL, Marchwicki RH, Strano AJ. Recovery and characterization of a coronavirus from military dogs with diarrhea. Proc Annu Meet U S Anim Health Assoc. 1974: 359-66.
- Bolles M, Donaldson E, Baric R. SARS-CoV and emergent coronaviruses: viral determinants of interspecies transmission. Curr Opin Virol. 2011; 1: 624-34.
- Brownlie J, Sibley D. What can animal coronaviruses tell us about emerging human coronaviruses? Vet Record April 2020:18/25.
- Bucknall RA, King LM, Kapikian AZ, Chanock RM. Studies with human coronaviruses. II. Some properties of strains 229E and OC43. Proc Soc Exp Biol Med. 1972; 139: 722-7. Int. J. Biol. Sci. 2020, Vol. 16
- Calisher CH, Childs JE, Field HE, Holmes KV, Schountz T. Bats: important reservoir hosts of emerging viruses. Clin Microbiol Rev. 2006; 19: 531-45.
- CDC, Coronavirus Disease 2019 (COVID-19) - pets & other animals (consulta: 01/11/2020). https://www.cdc.gov/coronavirus/2019-ncov/animals/pets-other-animals.html
- Centers for Disease C, Prevention. Prevalence of IgG antibody to SARS-associated coronavirus in animal traders--Guangdong Province, China, 2003. MMWR Morb Mortal Wkly Rep. 2003; 52: 986-7.
- Chan JF, To KK, Tse H, Jin DY, Yuen KY. Interspecies transmission and emergence of novel viruses: lessons from bats and birds. Trends Microbiol. 2013; 21: 544-55.
- Cheng VC, Lau SK, Woo PC, Yuen KY. Severe acute respiratory syndrome coronavirus as an agent of emerging and reemerging infection. Clin Microbiol Rev. 2007; 20: 660-94.
- Coleman CM, Frieman MB. Emergence of the Middle East respiratory syndrome coronavirus. PLoS Pathog. 2013; 9: e1003595.
- Corman VM, Baldwin HJ, Tateno AF, Zerbinati RM, Annan A, Owusu M, et al. Evidence for an Ancestral Association of Human Coronavirus 229E with Bats. J Virol. 2015; 89: 11858-70.
- Crossley BM, Mock RE, Callison SA, Hietala SK. Identification and characterization of a novel alpaca respiratory coronavirus most closely related to the human coronavirus 229E. Viruses. 2012; 4: 3689-700.
- Cui J, Li F, Shi ZL. Origin and evolution of pathogenic coronaviruses. Nat Rev Microbiol. 2019; 17: 181-92.
- Cunningham CH, Stuart HO. Cultivation of the virus of infectious bronchitis of chickens in embryonated chicken eggs. Am J Vet Res. 1947; 8: 209-12.
- Domańska-Blicharz K, Woźniakowski G, Konopka B, et al. Animal Coronaviruses in the Light of COVID-19. J Vet Res. 2020; 64(3):333-345.
- Duffy S. Why are RNA virus mutation rates so damn high? PLoS Biol. 2018; 16: e3000003.
- ECDC, 2020. Detection of new SARS-CoV-2 variants related to. Mink. Rapid risk assessment.
- Evermann JF, Baumgartener L, Ott RL, Davis EV, McKeirnan AJ. Characterization of a feline infectious peritonitis virus isolate. Vet Pathol. 1981; 18: 256-65.
- Forni D, Cagliani R, Clerici M, Sironi M. Molecular Evolution of Human Coronavirus Genomes. Trends Microbiol. 2017; 25: 35-48.
- Gao H, Yao H, Yang S, Li L. From SARS to MERS: evidence and speculation. Front Med. 2016; 10: 377-82.
- Graham RL, Baric RS. Recombination, reservoirs, and the modular spike: mechanisms of coronavirus cross-species transmission. J Virol. 2010; 84: 3134-46.
- Guan Y, Zheng BJ, He YQ, Liu XL, Zhuang ZX, Cheung CL, et al. Isolation and characterization of viruses related to the SARS coronavirus from animals in southern China. Science. 2003; 302: 276-8.
- Hilgenfeld R, Peiris M. From SARS to MERS: 10 years of research on highly pathogenic human coronaviruses. Antiviral Res. 2013; 100: 286-95.
- Hofmann H, Pyrc K, van der Hoek L, Geier M, Berkhout B, Pohlmann S. Human coronavirus NL63 employs the severe acute respiratory syndrome coronavirus receptor for cellular entry. Proc Natl Acad Sci U S A. 2005; 102: 7988-93.
- Hon CC, Lam TY, Shi ZL, Drummond AJ, Yip CW, Zeng F, et al. Evidence of the recombinant origin of a bat severe acute respiratory syndrome (SARS)-like coronavirus and its implications on the direct ancestor of SARS coronavirus. J Virol. 2008; 82: 1819-26.
- Hu B, Ge X, Wang LF, Shi Z. Bat origin of human coronaviruses. Virol J. 2015;12: 221.
- Hu B, Guo H, Zhou P, Shi ZL. Characteristics of SARS-CoV-2 and COVID-19 [published online ahead of print, 2020 Oct 6]. Nat Rev Microbiol. 2020;1-14.
- Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020.
- Huynh J, Li S, Yount B, Smith A, Sturges L, Olsen JC, et al. Evidence supporting a zoonotic origin of human coronavirus strain NL63. J Virol. 2012;86: 12816-25.
- Jan Molenaar, Robert. et al. (2020). Clinical and Pathological Findings in SARS-CoV-2 Disease Outbreaks in Farmed Mink (Neovison vison). Vet Pathol. 2020 Sep;57(5): 653-657.doi: 10.1177/0300985820943535. Epub 2020 Jul 14.
- Jianzhong et al. (2020). Susceptibility of ferrets, cats, dogs, and other domesticated animals to SARS-coronavirus 2. Science. 2020 May 29;368(6494):1016-1020. doi: 10.1126/science.abb 7015. Epub 2020 Apr 8.
- Kim et al. (2020). Infection and Rapid Transmission of SARS-CoV-2 in Ferrets. Cell Host & Microbe 27, 704–709 May 13, 2020 a 2020 Elsevier Inc. https://doi.org/10.1016/j.chom.2020.03.023
- Lam T, Shum M, Zhu H, Tong Y, Ni X, Liao Y, et al. Identification of 2019-nCoV related coronaviruses in Malayan pangolins in southern China. BIORXIV. 2020; doi: 10.1101/2020.02.13.945485
- Lau and Chan. Coronaviruses: emerging and re-emerging pathogens in humans and animals. Virology Journal. 2015; 12:209.
- Lau SK, Woo PC, Li KS, Huang Y, Tsoi HW, Wong BH, et al. Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats. Proc Natl Acad Sci U S A. 2005; 102: 14040-5.
- Lau SKP, Zhang L, Luk HKH, Xiong L, Peng X, Li KSM, et al. Receptor Usage of a Novel Bat Lineage C Betacoronavirus Reveals Evolution of Middle East Respiratory Syndrome-Related Coronavirus Spike Proteins for Human Dipeptidyl Peptidase 4 Binding. J Infect Dis. 2018; 218: 197-207.
- Lee KM, Moro M, Baker JA. Transmissible gastroenteritis in pigs. Am J Vet Res. 1954; 15: 364-72.
- Li W, Shi Z, Yu M, Ren W, Smith C, Epstein JH, et al. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005; 310: 676-9.
- Liu P, Chen W, Chen JP. Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica). Viruses. 2019;11.
- Luo CM, Wang N, Yang XL, Liu HZ, Zhang W, Li B, et al. Discovery of Novel Bat Coronaviruses in South China That Use the Same Receptor as Middle East Respiratory Syndrome Coronavirus. J Virol. 2018; 92.
- Menachery VD, Graham RL, Baric RS. Jumping species-a mechanism for coronavirus persistence and survival. Curr Opin Virol. 2017; 23: 1-7.
- OIE (World Organization for Animal Health). Guidance on working with farmed animals of species susceptible to infection with SARS-CoV-2. Draft published 05/11/2020.
- OIE. Consideraciones para el muestreo, las pruebas y la notificación de SARS-CoV-2 en animales (consulta: 01/11/2020). https://www.oie.int/fileadmin/Home/esp/Our_scientific_expertise/docs/pdf/COV-19/E_Sampling_Testing_and_Reporting_of_SARS-CoV-2_in_animals_final_7May_2020.pdf
- OIE. Ficha técnica. https://www.oie.int/app/uploads/2021/05/es-factsheet-sars-cov-2.pdf
- OIE. Preguntas y respuestas sobre Covid-19. https://www.oie.int/es/nuestra-experiencia-cientifica/informaciones-especificas-y-recomendaciones/preguntas-y-respuestas-del-nuevo-coronavirus-2019/
- Oreshkova, N. et al. (2020). SARS-CoV-2 infection in farmed minks, the Netherlands, April and May 2020. Eurosurveillance. Jun; 25 (23): 2001005.doi:10.2807/1560-7917.ES.2020.25.23.2001005.
- Organización Colegial Veterinaria. Vídeo sobre Covid-19, animales de compañía y medidas de protección. https://www.youtube.com/watch?v=nl80FC3pxEw
- Oude Munnink, B. B. et al. (2020). Transmission of SARS-CoV-2 on mink farms between humans and mink and back to humans. Science 10.1126/science.abe5901.
- Poon LL, Chu DK, Chan KH, Wong OK, Ellis TM, Leung YH, et al. Identification of a novel coronavirus in bats. J Virol. 2005; 79: 2001-9.
- Poudel U, Subedi D, Pantha S, Dhakal S. Open Veterinary Journal, (2020), Vol. 10(3): 239–251.
- Reusken CB, Haagmans BL, Muller MA, Gutierrez C, Godeke GJ, Meyer B, et al. Middle East respiratory syndrome coronavirus neutralising serum antibodies in dromedary camels: a comparative serological study. Lancet Infect Dis. 2013; 13: 859-66.
- Reusken, C.B. et al. (2020). Rapid assessment of regional SARS-CoV-2 community transmission through a convenience sample of healthcare workers, the Netherlands, March 2020. Eurosurveillance. 2020 Mar; 25 (12): 2000334.doi: 10.2807/1560-7917.ES.2020.25.12.2000334.
- Sabir JS, Lam TT, Ahmed MM, Li L, Shen Y, Abo-Aba SE, et al. Co-circulation of three camel coronavirus species and recombination of MERS-CoVs in Saudi Arabia. Science. 2016; 351: 81-4.
- SARS-CoV-2 in animals. (2020). Document prepared by the Animal Virology Coordination Team of MediLabSecure. Animal Haelth Research Center. INIA-CISA. Valdeolmos, Madrid, Spain.
- Shen Min et al. (2020). SARS-Cov-2 Infection of Cats and Dogs. Prepints.
- Statens Serum Institut. Risikovurdering af human sundhed ved fortsat minkavl. 3 November 2020. Copenhagen: Statens Serum Institut; 2020.
- Su S, Wong G, Shi W, Liu J, Lai ACK, Zhou J, et al. Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses. Trends Microbiol. 2016;24: 490-502.
- Tao Y, Shi M, Chommanard C, Queen K, Zhang J, Markotter W, et al. Surveillance of Bat Coronaviruses in Kenya Identifies Relatives of Human Coronaviruses NL63 and 229E and Their Recombination History. J Virol. 2017;91.
- Temmam, S. et al. (2020). Absence of SARS-CoV-2 infection in cats and dogs in close contact with a cluster of COVID-19 patients in a veterinary campus. One Health. 2020 Dec;10:100164. doi: 10.1016/j.onehlt.2020.100164. Epub 2020 Aug 29.
- To KK, Hung IF, Chan JF, Yuen KY. From SARS coronavirus to novel animal and human coronaviruses. J Thorac Dis. 2013; 5 Suppl 2: S103-8.
- Tu C, Crameri G, Kong X, Chen J, Sun Y, Yu M, et al. Antibodies to SARS coronavirus in civets. Emerg Infect Dis. 2004; 10: 2244-8.
- Vincent C. C. Cheng, Susanna K. P. Lau, Patrick C. Y. Woo, Kwok Yung Yuen. (2007). Severe Acute Respiratory Syndrome Coronavirus as an Agent of Emerging and Reemerging Infection. Clinical Microbiology Reviews. Vol. 20 (4): 660-694.
- Weiss SR, Navas-Martin S. Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus. Microbiol Mol Biol Rev. 2005; 69: 635-64.
- Woo PC, Lau SK, Huang Y, Yuen KY. Coronavirus diversity, phylogeny and interspecies jumping. Exp Biol Med (Maywood). 2009; 234: 1117-27.
- Woo PC, Lau SK, Tsoi HW, Huang Y, Poon RW, Chu CM, et al. Clinical and molecular epidemiological features of coronavirus HKU1-associated community-acquired pneumonia. J Infect Dis. 2005; 192: 1898-907.
- World Organisation for Animal Health. COVID-19 Portal. Events in animals [cited 11 November 2020]. Available from: https://www.oie.int/en/scientific-expertise/specific-information-and-recommendations/questions-and-answers-on-2019novelcoronavirus/events-in-animals/
- Zaki AM, van Boheemen S, Bestebroer TM, Osterhaus AD, Fouchier RA. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med. 2012; 367: 1814-20.
- Zhang G, Cowled C, Shi Z, Huang Z, Bishop-Lilly KA, Fang X, et al. Comparative analysis of bat genomes provides insight into the evolution of flight and immunity. Science. 2013; 339: 456-60.
- Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020.
 by Multimédica
by Multimédica